
Inhoudsopgave:
- Stap 1: Zoek MRI- en MRA-scans van een normaal brein en een brein dat is aangetast door Moyamoya
- Stap 2: Laad afbeeldingen in MATLAB en wijs afbeeldingen toe aan een variabele om afbeeldingen weer te geven
- Stap 3: Verbeter langwerpige structuren in intensiteitsbeeld met filtering op meerdere schalen
- Stap 4: Voer een 2D-mediaanfilter uit
- Stap 5: Maskeer de afbeelding
- Stap 6: Selecteer de MRA-scans voor statistische tests
- Stap 7: Bereken het gebied van bloedvaten ter voorbereiding op statistische tests
- Stap 8: Voer een Independent Samples T-test uit
- Auteur John Day day@howwhatproduce.com.
- Public 2024-01-30 11:18.
- Laatst gewijzigd 2025-01-23 15:01.

Moyamoya, "rookwolk", is een zeldzame ziekte die wordt veroorzaakt door de verstopping van slagaders in de basale ganglia, een gebied aan de basis van de hersenen. De ziekte is een progressieve cerebrovasculaire ziekte die vooral kinderen treft. Symptomen van moyamoya zijn onder meer een eerste beroerte, constante mini-beroertes, spierzwakte, verlamming of toevallen als gevolg van de progressieve vernauwing van de slagaders. Zonder behandeling veroorzaakt moyamoya problemen met spraak, sensorische stoornissen en beschadigd bewustzijn. In ons project zullen we MATLAB gebruiken om een MRI- of MRA-beeld voor te bewerken met behulp van verschillende filters om ruis in het beeld te verminderen om het getroffen gebied te lokaliseren. Daarnaast zullen we een functieverbetering gebruiken om de getroffen gebieden nauwkeuriger te lokaliseren. Bovendien zullen we dan een onafhankelijke t-test uitvoeren om te bepalen of er een significant verschil is tussen de hoeveelheid bloedvaten in een normaal brein in vergelijking met een brein dat is aangetast door moyamoya.
Stap 1: Zoek MRI- en MRA-scans van een normaal brein en een brein dat is aangetast door Moyamoya




Deze afbeeldingen zijn de scans die we hebben gebruikt voor het project dat we online hebben gevonden. De twee afbeeldingen met de bloedvaten in het midden zijn MRA-scans, terwijl de andere twee afbeeldingen MRI-scans zijn.
De volgende links zijn waar deze afbeeldingen te vinden zijn:
static.cambridge.org/resource/id/urn:cambr…
static.cambridge.org/resource/id/urn:cambr…
Stap 2: Laad afbeeldingen in MATLAB en wijs afbeeldingen toe aan een variabele om afbeeldingen weer te geven

Om het proces te starten, begint u met het wissen van het opdrachtvenster, sluit u alle mogelijke figuren en grafieken die mogelijk al open zijn en wist u de variabelen die al in uw werkruimte zijn toegewezen.
Maak daarna een for-lus van 1 tot 2 met het commando i = [1:2]
Laad daarna de MRA-afbeeldingen met behulp van de opdracht imread(sprintf('filename%.filetype', i)) om de afbeeldingen te lezen uit de bestanden die zijn gespecificeerd door de bestandsnaam, gevolgd door het nummer dat wordt gebruikt voor de lus met sprintf voor het laden van batches en toewijzen het naar een variabele.
Om de afbeelding vervolgens in een figuur weer te geven, gebruikt u het commando imshow(I).
Gebruik het commando colormap(grijs) om een grijze kleurenkaart toe te wijzen.
Om kleur volledig te elimineren en de 3D-matrix voor de afbeeldingen om te zetten in 2D, gebruikt u de opdracht rgb2gray(I) en wijst u deze toe aan een afzonderlijke variabele.
Laad vervolgens de MRI-beelden met behulp van de eerder genoemde opdracht of imread(sprintf('filename%.filetype', i)) en wijs deze toe aan een nieuwe variabele
Herhaal de opdracht rgb2gray met de nieuwe variabele die voor de MRI-beelden wordt gebruikt.
Indien nodig kunt u het formaat van een afbeelding wijzigen met het commando imresize(A, scale) en toewijzen aan een aparte variabele.
Stap 3: Verbeter langwerpige structuren in intensiteitsbeeld met filtering op meerdere schalen

Gebruik een nieuwe variabele en gebruik het commando fibermetric(A) om de buisvormige structuren in de afbeeldingen te verbeteren
Gebruik bij de vorige variabele de opdracht histeq(B) om de histogram-egalisatie te verbeteren door de intensiteit van de afbeeldingen te transformeren en toe te wijzen aan een nieuwe variabele.
Geef het histogram weer met het commando imhist(B)
Maak een nieuwe variabele om een drempel voor het filter te maken. Wijs in dit geval de vorige variabele> 0.875 toe, waarbij de pixelintensiteit onder de waarde van 0.875. wordt uitgefilterd
Maak daarna een nieuwe afbeelding en gebruik de opdracht imshow(A) om de nieuwe gefilterde afbeelding weer te geven.
Stap 4: Voer een 2D-mediaanfilter uit

Voer met de opdracht medfilt2(A, [m n]) een 2D-mediaanfilter uit, waarbij elke uitvoerpixel de mediaanwaarde in de mxn-grens rond de respectieve pixel in de invoerafbeelding bevat.
Maak een nieuwe figuur en gebruik imshow(A) om de mediaan gefilterde afbeelding weer te geven.
Stap 5: Maskeer de afbeelding

Gebruik de mediaan gefilterde afbeelding en gebruik de opdracht [labeledImage, numberOfBlots] = bwlabel(A) om het aantal witte vlekken in de afbeelding te tellen
Gebruik vervolgens de region props functie states = regionprops(labeledImage, 'Area') om de oppervlakten van elke vlek of bloedvat te berekenen
Wijs alle gebieden toe aan één variabele
Gebruik vervolgens een andere variabele en tel het aantal blots dat groter is dan 50 pixels
Sorteer daarna alle blots die kleiner zijn dan 50 pixels in aflopende volgorde met het commando [sortedAreas, SortIndicies] = sort(Areas, 'descend')
Gebruik vervolgens een andere variabele en gebruik het commando ismember(labeledImage, SortIndicies(1:numberToExtract)) om een array te retourneren met elementen van labeledImage worden gevonden in gesorteerdIndicies van nummer 1 tot het aantal bloedvaten om een logische 1 (true) te retourneren. een logische 0 (false).
Zoek met de variabele in de vorige stap de punten die waar zijn (waarden > 0) en maak een logische array om een binaire afbeelding te maken en deze toe te wijzen aan een nieuwe variabele.
Maak een nieuwe figuur en gebruik imshow(A) de nieuwe binaire afbeelding.
Keer vervolgens de afbeelding om met de opdracht imcomplement(A) en wijs deze toe aan een andere variabele.
Om een gemaskeerde afbeelding te maken, gebruikt u een nieuwe variabele met het commando resizedimage.*uint8(invertedimage)
Maak een nieuwe figuur en gebruik imshow(A) om de gemaskeerde afbeelding weer te geven.
Om de hele code te beëindigen, moet je het commando 'end' gebruiken om de hele for-lus te beëindigen
Stap 6: Selecteer de MRA-scans voor statistische tests



Selecteer ter voorbereiding op statistische tests de MRA-scans die moeten worden gebruikt voor de t-test voor onafhankelijke steekproeven. Omdat onze twee monsters Moyamoya-hersenen zullen zijn en normale hersenen, selecteert u een behoorlijk aantal MRA-scans van elke groep.
Stap 7: Bereken het gebied van bloedvaten ter voorbereiding op statistische tests

De statistische test zal zich richten op de lengte of hoeveelheid bloedvaten die op de MRA-scans worden getoond. We moeten dus het gebied van de bloedvaten berekenen voordat we het vergelijken.
Begin met het filteren van de MRA's van normale hersenen en het berekenen van de hoeveelheid bloedvaten. Voer hiervoor een for-lus uit. Omdat er drie afbeeldingen zijn, is de voorwaarde i = [1:3].
Open de afbeelding met de opdracht imread en wijs deze toe aan een variabele.
Maak vervolgens een if/els-statement met het if, else-commando. Gebruik voor de if-instructie de opdracht size(A, 3)==3, waarbij A de variabele is die wordt gebruikt om de afbeelding te openen, om een if-instructie te maken voor wanneer de derde dimensie van de array 3 is. Converteer vervolgens de afbeelding naar 2D en verwijder kleur met het commando rgb2gray(A) en wijs het toe aan een nieuwe variabele. Gebruik het commando imresize(A, [m n]) om het formaat van de afbeelding te wijzigen. In dit geval hebben we het formaat van de afbeeldingen aangepast naar 1024 x 1024 matrix. Om de buisvormige structuren van de afbeelding te verbeteren, gebruikt u opnieuw de opdracht fibermetric en wijst u deze toe aan een nieuwe variabele.
Het volgende is voor de else-instructie. Als de afbeelding geen 3D-matrix is, willen we de conversie overslaan. Doe hetzelfde als het if-statement, maar zonder het rgb2gray(A)-commando.
Maak een nieuwe variabele en stel deze gelijk in aan de variabele van de vezelmetrische stap groter dan 0,15. Dit drempelt het beeld voor intensiteiten groter dan 0,15.
We herhalen de regels met codes uit de stappen 4 en 5 van de instructable van de mediaanfilterlijn tot de imshow (I) -regel. Gebruik daarna het commando sum(I(:)) om alle pixels die bloedvaten vormen bij elkaar op te tellen en toe te wijzen aan een aparte variabele. Geef een nieuwe variabele de naam NormalBloodVessels(i) en stel deze in op de variabele van de opdracht sum(I(:)). Dit voegt de gegevens toe aan de matrix.
Beëindig de lus en herhaal, behalve voor de MRA's van hersenen die zijn aangetast door Moyamoya. Noem de variabele aan het einde MoyaMoyaBloodVessels(i) om deze niet te verwarren met de normale hersen-MRA's.
Stap 8: Voer een Independent Samples T-test uit

Aangezien er twee onafhankelijke steekproeven en een kleine populatie zijn, voert u een t-toets voor onafhankelijke steekproeven uit.
Maak en noem een functie die een onafhankelijke t-test uitvoert om te bepalen of het aantal bloedvaten in de MRA's van normale hersenen significant gelijk is aan die van MRA's van hersenen die zijn aangetast door Moyamoya.
Geef de hypothese voor de test weer met het commando disp('X'). Geef op de eerste regel 'Hypothesen voor twee steekproeven t-test' weer. Geef op de tweede regel weer: "H0 = Het aantal bloedvaten van een normaal brein is gelijk aan het aantal bloedvaten van een brein met de ziekte van Moyamoya", om de nulhypothese te formuleren. Geef op de derde regel weer: "HA = Het aantal bloedvaten van een normaal brein is niet gelijk aan het aantal bloedvaten van een brein met de ziekte van Moyamoya." om de alternatieve hypothese te formuleren.
Bereken met behulp van een 95%-betrouwbaarheidsinterval en een steekproefomvang van 3 de t-score met het commando tinv([0.025 0.975], 2) en wijs het toe aan variabele t. Gebruik de opdracht var(NormalBloodVessels) en var(MoyaMoyaBloodVessels) en wijs ze toe aan variabelen om de varianties van beide datasets te berekenen.
Controleer of de varianties bijna gelijk zijn of niet. Doe dit door een if/els-statement te maken met het if, else-commando. Voor de voorwaarde in het if-statement, schrijf A / B == [0.25:4], waarbij A de variabele is die de variantie van normale bloedvaten verklaart en B de variabele is die de variantie van Moyamoya-bloedvaten verklaart. 0,25 en 4 komen uit een algemene schatting om te bepalen of varianties gelijk zijn of niet. Voer vervolgens de t-test met twee steekproeven uit met [h, p] = ttest2(A, B, 0,05, 'beide', 'gelijk'), waarbij A en B dezelfde variabelen zijn als eerder vermeld. Gebruik voor de else-instructie [h, p] = ttest2(A, B, 0.05, 'beide', 'onequal') om een t-test met twee steekproeven uit te voeren in het geval dat de varianties niet gelijk zijn. Beëindig het if/else statement. Dit berekent p.
Maak een if/else-statement dat een conclusie toont op basis van de waarde van p. De voorwaarde voor het if-statement is p > 0,05. Aangezien we de nulhypothese doorgaans niet verwerpen wanneer de waarde van p groter is dan 0,05, gebruikt u de opdracht disp('X') om "Omdat de p-waarde groter is dan 0,05, kunnen we de nulhypothese niet verwerpen", en " Daarom kunnen we niet ontkennen dat de hoeveelheid bloedvaten van een normaal brein gelijk is aan dat van een brein met de ziekte van Moyamoya." In de else-instructie, aangezien we de nulhypothese doorgaans verwerpen wanneer de waarde van p lager is dan 0,05, gebruikt u de opdracht disp('X') om weer te geven "Omdat de p-waarde kleiner is dan 0,05, verwerpen we de nulhypothese," en " Daarom kunnen we niet ontkennen dat de hoeveelheid bloedvaten van een normaal brein niet gelijk is aan dat van een brein met de ziekte van Moyamoya." Beëindig het if/else statement.
Aanbevolen:
Game Design in Flick in 5 stappen: 5 stappen

Game-ontwerp in Flick in 5 stappen: Flick is een heel eenvoudige manier om een game te maken, vooral zoiets als een puzzel, visuele roman of avonturengame
Gezichtsdetectie op Raspberry Pi 4B in 3 stappen: 3 stappen

Gezichtsdetectie op Raspberry Pi 4B in 3 stappen: In deze Instructable gaan we gezichtsdetectie uitvoeren op Raspberry Pi 4 met Shunya O/S met behulp van de Shunyaface-bibliotheek. Shunyaface is een bibliotheek voor gezichtsherkenning/detectie. Het project streeft naar de hoogste detectie- en herkenningssnelheid met
Doe-het-zelfspiegel in eenvoudige stappen (met LED-stripverlichting): 4 stappen

DIY make-upspiegel in eenvoudige stappen (met behulp van LED-stripverlichting): In dit bericht heb ik een doe-het-zelfspiegel gemaakt met behulp van de LED-strips. Het is echt gaaf en je moet ze ook proberen
Hoe plug-ins in WordPress te installeren in 3 stappen: 3 stappen
Hoe plug-ins in WordPress te installeren in 3 stappen: In deze tutorial laat ik je de essentiële stappen zien om de WordPress-plug-in op je website te installeren. In principe kunt u plug-ins op twee verschillende manieren installeren. De eerste methode is via ftp of via cpanel. Maar ik zal het niet opsommen, want het is echt compl
Akoestische levitatie met Arduino Uno stap voor stap (8 stappen): 8 stappen
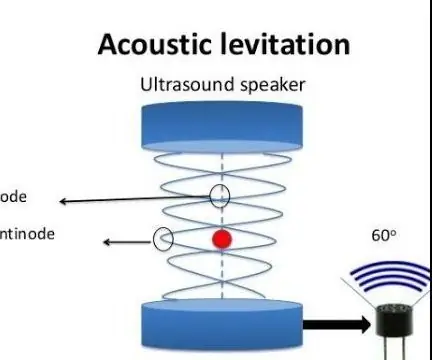
Akoestische levitatie met Arduino Uno Stap voor stap (8-stappen): ultrasone geluidstransducers L298N Vrouwelijke DC-adapter voeding met een mannelijke DC-pin Arduino UNOBreadboardHoe dit werkt: eerst upload je code naar Arduino Uno (het is een microcontroller uitgerust met digitale en analoge poorten om code te converteren (C++)
