Inhoudsopgave:
- Auteur John Day day@howwhatproduce.com.
- Public 2024-01-30 11:17.
- Laatst gewijzigd 2025-01-23 15:01.
Door: Madhumita Kannan, Henry Nguyen, Ashley Urrutia Avila, Mei Jin
Deze MATLAB-code is een programma om de exacte grootte, vorm en locatie te detecteren van een tumor die wordt gevonden in MRI-scans van de hersenen van een patiënt. Dit programma is oorspronkelijk ontworpen om te werken met tumordetectie in MRI-scans van de hersenen, maar kan ook worden gebruikt voor kankerdiagnostiek in andere orgaanscans.
De volgende instructies beschrijven eerst de methoden voor beeldanalyse door middel van filteren en opschonen van de MRI-scan, door binariseren, mediaan filteren en schuifvensters. Vervolgens zal het instrueren hoe de tumor te isoleren met behulp van een vooraf gegenereerd elliptisch masker en het verder te filteren om de omtrek van de vorm van de tumor te schetsen.
Zodra de tumor is gedetecteerd, wordt in de instructies verder beschreven hoe u dit programma in een grafische gebruikersinterface (GUI) kunt opnemen. Doorheen deze instructies zullen de juiste code en bestanden worden bijgevoegd om uit te leggen hoe deze MRI-scananalyse werkt.
Enkele dingen om te weten, te downloaden en klaar te hebben voordat u doorgaat met deze instructable: 1. Zorg ervoor dat u de nieuwste versie van MATLAB hebt gedownload. U kunt R2018b hier installeren:
2. Om dit programma uit te voeren, moet u toegang hebben tot MRI-hersenscanbestanden. Hoewel sommige altijd te vinden zijn op Google-afbeeldingen, kan een grondige en nauwkeurige analyse worden uitgevoerd op basis van de juiste afbeeldingen van verschillende lagen hersenscans voor elke patiënt. U kunt vanuit deze database toegang krijgen tot de bestanden van 20 verschillende patiënten met glioblastoom voor en na de behandeling:
3. De focus van dit programma en de verschillende methoden die dit project begeleiden, worden beschreven in dit onderzoeksrapport:
Stap 1: Start de grafische gebruikersinterface (GUI)
De eerste stap zou zijn om de grafische gebruikersinterface, GUI, te maken en te starten. Dit kan worden gedaan door guide in het opdrachtvenster te typen, op enter te drukken en een nieuwe GUI te maken. Zodra deze stap is voltooid, kunt u beginnen met het maken van functies zoals assen, statische tekst, tekst bewerken en drukknoppen die worden weergegeven zodra het programma is uitgevoerd en waarmee de gebruiker kan communiceren. Deze functies kunnen worden bewerkt en gemanipuleerd via de eigenschappencontrole, maar de belangrijkste functie die moet worden gewijzigd bij het maken van deze functies is de tagnaam. Het is belangrijk om de tagnaam van elke geïmplementeerde functie te wijzigen, omdat dit ons in staat zal stellen een herkenbare callback-functie te creëren. Zodra u tevreden bent met de lay-out van uw GUI, kunt u doorgaan met het laden van de DICOM-bestanden die in de GUI worden weergegeven.
Stap 2: MRI-afbeeldingen laden en lezen in MATLAB
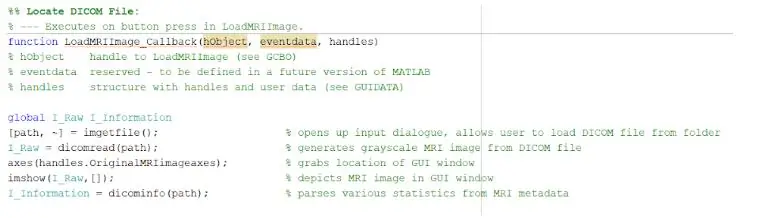
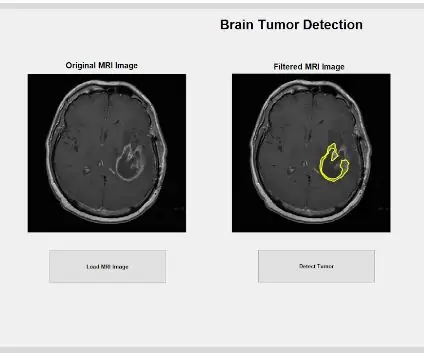
Om de DICOM-bestanden te laden, moet u de terugbelfunctie die wordt uitgevoerd wanneer u op de knop "MRI-afbeelding laden" drukt, correct initialiseren. Zodra dit is voltooid, moet u een globale variabele maken waarmee het beeld wordt weergegeven op de assen van de handvatten waar u het originele MRI-beeld wilt weergeven. De MRI-scanbeelden die uit de database zijn gedownload, zijn allemaal in DICOM-geformatteerde bestanden die in uw MATLAB-directory moeten worden geladen. Lokaliseer het bestand met behulp van imgetfile om ze in het programma te laden. De afbeeldingen worden gelezen met behulp van de ingebouwde MATLAB-functie 'dicomread', en de eerste onbewerkte afbeelding voor elk bestand wordt ingebed in de linker GUI-assen met behulp van imshow.
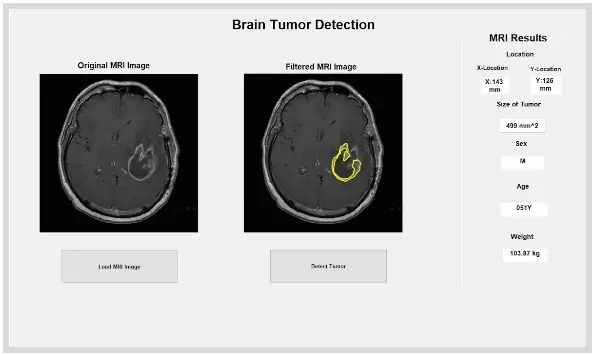
De ingebouwde MATLAB-functie 'dicominfo' is ook uiterst nuttig bij het adresseren van alle informatie van elk MRI-dicom-bestand. We hebben deze functie gebruikt om alle beschrijvende informatie van de patiënten te extraheren, zoals hun geslacht, leeftijd, gewicht en lengte. Deze functie geeft u ook de stapelvolgorde die handig is voor de implementatie van het programma binnen de grafische gebruikersinterface. We hebben variabelen gemaakt voor elk van de beschrijvende informatie van de patiënten die zullen worden gebruikt voor de GUI wanneer de detectieknop wordt ingedrukt.
Stap 3: Beeldfiltering

Nadat het DICOM-bestand van de onbewerkte afbeelding is geladen en gelezen, moet de afbeelding worden geconverteerd van grijswaarden naar een binaire vorm die alleen uit zwarte en witte pixels bestaat. We gebruikten de functie 'imbinariseren' om een binaire afbeelding van de onbewerkte afbeelding te maken door het beheersen van aspecten van adaptieve drempelwaarde bij een gevoeligheidswaarde van 0,59. De standaard drempelgevoeligheidsfactor, 0,5 was laag en kon de helderdere vlekken en vlekken van de afbeelding niet detecteren, dus hebben we deze verhoogd naar 0,59.
Het gebinariseerde beeld wordt vervolgens verwerkt door een mediaanfilter met behulp van de functie 'medfilt2' omdat het gebinariseerde beeld tweedimensionaal is. We hebben elke uitvoerpixel zo ingesteld dat deze de mediaanwaarde in de 5 x 5 buurt rond de overeenkomstige pixel in de binaire invoerafbeelding bevat. Dit vermindert de ruis en behoudt de randen in een vierkant van 5 x 5 rond elke pixel. Vervolgens passen we een schuifvenster toe met behulp van 'strel' om een schijfvormig plat structurerend element te creëren met een buurtradius van 2 om elke centrale oorsprongpixel in elke schijfomgeving te identificeren. We hebben een schijfstructureringselement gebruikt omdat we elke cirkelvormige plek en de pixels binnen elke plek analyseren, dus een schijfvormelement is nuttiger.
Nadat de afbeelding is gefilterd, kan deze worden schoongemaakt met behulp van de 'imclose'-functie om de zwarte vlekken tussen de gefilterde witte pixels in de afbeelding te verwijderen en alle gaten eromheen te sluiten. De volledig bewerkte afbeelding kan vervolgens worden uitgezet in de tweede subplot van de vooraf toegewezen afbeelding, waardoor een vergelijking tussen de onbewerkte en gefilterde afbeelding mogelijk is.
Stap 4: Tumorisolatie door elliptisch masker


De heldere vlekken van de tumor kunnen vervolgens worden geïsoleerd van het gefilterde hoofdbeeld via een vooraf gegenereerd elliptisch masker. Om dit masker te maken, moet u de grootte van het originele, onbewerkte MRI-scanbeeld kennen en de rij- en kolomlengte gebruiken, respectievelijk als x- en y-coördinaten, om de middelste coördinaten voor de elliptische trainer toe te wijzen. We stellen de y-as in als hoofdas met een straal van 50 eenheden vanaf het centrum en de secundaire as als een straal van 40 eenheden vanaf het centrum.
We gebruikten de MATLAB-functie 'meshgrid' om een cartesiaans vlak te genereren met tweedimensionale rastercoördinaten op basis van de coördinaten in vectoren van 1 tot de lengte van de x-as, en van 1 tot de lengte van de y-as van de afbeelding. Col is een matrix waarbij elke rij een kopie is van de x-as, en Rij is een matrix waarbij elke kolom een kopie is van de y-as. Het cartesiaanse raster dat wordt weergegeven door de coördinaten Col en Row heeft lengte (1:Y_Size) rijen en lengte (1:X_Size) kolommen. Gebruik de indices van Col en Row die zijn gegenereerd door het cartesiaanse raster om de ellipsvergelijking te bepalen, afhankelijk van de vooraf bepaalde straal- en middencoördinaten. De elliptische omtrek kan nu worden gevuld met de witte pixels die zijn gevonden op tumorvlekken.
Met behulp van het vooraf gegenereerde elliptische masker kunnen we de specifieke tumor die u wilt analyseren uit het gefilterde beeld verwijderen. Het elliptische masker detecteert welke plekken logisch binnen de omtrek van de ellips passen en accepteert dit als een plek op het gefilterde beeld om als tumor acceptabel te zijn. De functie 'bwareafilt' filtert vervolgens alle andere objecten buiten deze gedetecteerde tumor uit het beeld. We hebben empirisch een specifiek venster van 500 bij 4000 gebruikt op basis van de afmetingen van alle afbeeldingen. Vervolgens pasten we een ander schuifvenster toe met 'strel' als een plat schijfvormig structurerend element met een grotere buurtstraal van 6, om de openingen tussen elke centrale witte pixel in de gedetecteerde tumor te dichten. De gedetecteerde tumorplek wordt verder opgeschoond met behulp van 'imclose' om de zwarte pixels verder te elimineren en alle gaten op te vullen met 'imfill'. Deze bewerkte tumor kan vervolgens worden weergegeven in de derde subplot in de vooraf toegewezen plot om een vergelijking te maken tussen de geïsoleerde tumor en de originele en gefilterde beelden van de MRI-scan.
Stap 5: Tumoroverzicht
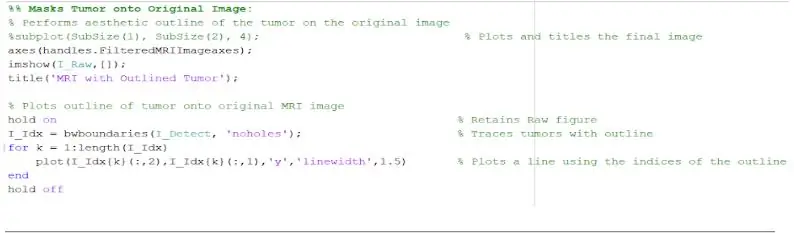
Nu de tumor is geïsoleerd met het masker, kan deze worden omlijnd en weergegeven op de originele afbeelding om de exacte locatie te tonen. Hiervoor hebben we de functie ‘bwboundaries’ gebruikt om de eerder gedetecteerde tumor met een omlijning op te sporen. We hebben de omtrek gespecificeerd om de gaten in het tumorobject niet op te nemen zoals het wordt omlijnd. Dit kan worden uitgezet op het originele, onbewerkte beeld, met behulp van een 'for'-lus die de omtrek rond de tumor uitzet met behulp van de indices van de lijn met een lijnbreedte van 1,5 pixels. Deze omtrek wordt vervolgens uitgezet op de onbewerkte afbeelding, waarbij de exacte grootte en locatie van de tumor wordt weergegeven, ten opzichte van de originele MRI-scan.
Stap 6: Analyse van de fysieke eigenschappen van tumoren
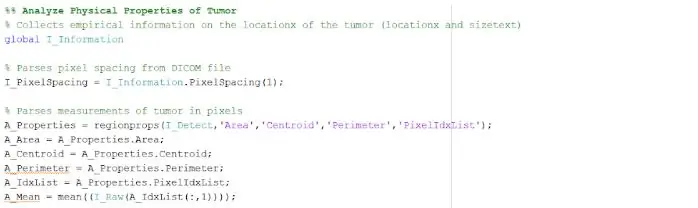
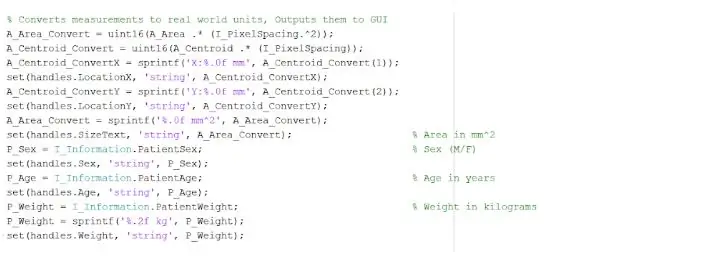
De geïsoleerde en omlijnde plek kan ons nuttige informatie geven over de grootte, het gebied en de locatie van de tumor. We gebruikten de functie 'regionprops' om de eigenschappen van de tumor te detecteren die betrekking hebben op gebied, omtrek, zwaartepunten en de pixelindexwaarde. Deze pixelindexwaarde geeft ons de echte wereldeenheden voor elke pixel van elke afbeelding, uniek voor elke scan. Deze eigenschappen kunnen vervolgens worden omgezet in real-world eenheden van millimeters. De empirische informatie die het programma ons geeft, is uniek voor elke MRI-scan en is uiterst nuttig bij het bepalen van de grootte, locatie en het type tumor, die de gebruikers kunnen analyseren en opnemen in de grafische gebruikersinterface.
Aanbevolen:
Digitale klok met netwerktijd met behulp van de ESP8266: 4 stappen (met afbeeldingen)

Digitale netwerkklok met de ESP8266: we leren hoe we een schattige kleine digitale klok kunnen bouwen die communiceert met NTP-servers en de netwerk- of internettijd weergeeft. We gebruiken de WeMos D1 mini om verbinding te maken met een wifi-netwerk, de NTP-tijd te verkrijgen en deze weer te geven op een OLED-module. De video hierboven
DIY slimme weegschaal met wekker (met wifi, ESP8266, Arduino IDE en Adafruit.io): 10 stappen (met afbeeldingen)

DIY Slimme Weegschaal Met Wekker (met Wi-Fi, ESP8266, Arduino IDE en Adafruit.io): In mijn vorige project ontwikkelde ik een slimme weegschaal met Wi-Fi. Het kan het gewicht van de gebruiker meten, het lokaal weergeven en naar de cloud sturen. U kunt hier meer informatie over krijgen op onderstaande link: https://www.instructables.com/id/Wi-Fi-Smart-Scale-wi
Draadloze afstandsbediening met 2,4 GHz NRF24L01-module met Arduino - Nrf24l01 4-kanaals / 6-kanaals zenderontvanger voor quadcopter - RC Helikopter - RC-vliegtuig met Arduino: 5 s
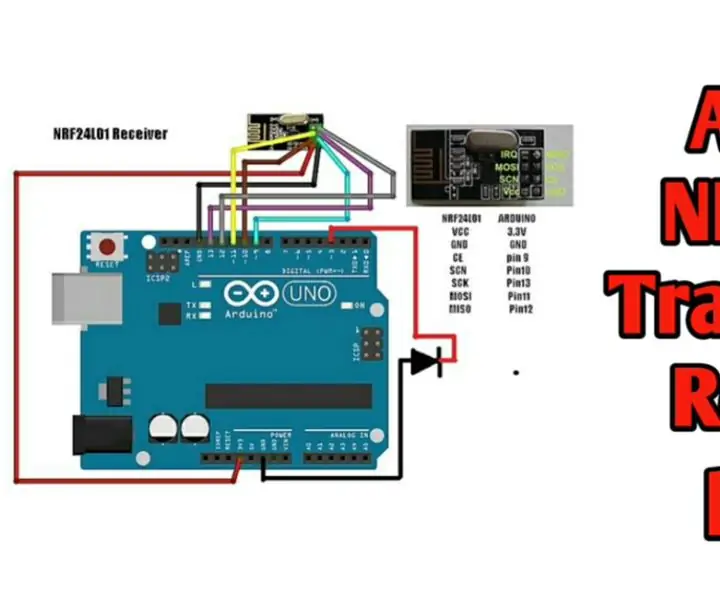
Draadloze afstandsbediening met 2,4 GHz NRF24L01-module met Arduino | Nrf24l01 4-kanaals / 6-kanaals zenderontvanger voor quadcopter | RC Helikopter | Rc-vliegtuig met Arduino: een Rc-auto besturen | Quadcopter | Drone | RC vliegtuig | RC-boot, we hebben altijd een ontvanger en zender nodig, stel dat we voor RC QUADCOPTER een 6-kanaals zender en ontvanger nodig hebben en dat type TX en RX is te duur, dus we gaan er een maken op onze
MATLAB App Designer gebruiken met Arduino: 5 stappen

MATLAB App Designer gebruiken met Arduino: MATLAB App Designer is een krachtige tool waarmee je grafische gebruikersinterfaces (GUI's) kunt ontwerpen met alle MATLAB-functionaliteiten. In deze tutorial gaan we een GUI maken om de helderheid van een LED te regelen via een eenvoudig te volgen stappen.N
Een gehoortest voor volwassenen maken met MATLAB: 6 stappen

Hoe u een gehoortest voor volwassenen maakt met MATLAB: DISCLAIMER: Onze test is GEEN medische diagnose en mag niet als zodanig worden gebruikt. Raadpleeg een medische professional om het gehoor nauwkeurig te meten. Met behulp van materialen die we al hadden, heeft onze groep een gehoortest gedaan. Onze test is alleen voor gebruik door volwassenen en tieners
