Inhoudsopgave:
- Stap 1: Vereisten
- Stap 2: Vereisten (vervolg)
- Stap 3: Vereisten (vervolg)
- Stap 4: Vereisten (vervolg)
- Stap 5: Matlab opruimen om u voor te bereiden op het uitvoeren van code
- Stap 6: Selecteer 10 normale oogbeelden en 10 beelden met diabetische retinopathiesymptomen
- Stap 7: Selecteer 10 normale oogbeelden en 10 beelden met diabetische retinopathiesymptomen (vervolg)
- Stap 8: Maak 2 variabelen (normaal en gediagnosticeerd) en stel ze elk gelijk aan 0
- Stap 9: Maak een for-lus om automatisch normale afbeeldingen te uploaden
- Stap 10: Maak een for-lus om automatisch normale afbeeldingen te uploaden (vervolg)
- Stap 11: Snijd de randen van de afbeelding bij
- Stap 12: Maak een grijsschaalafbeelding
- Stap 13: Maak een contrasterende afbeelding
- Stap 14: Verbeter het contrastbeeld
- Stap 15: Maak een gemiddeld filter
- Stap 16: Combineer het middelingsfilter met de contrasterende afbeelding
- Stap 17: Maak een nieuw gemiddeld masker door pixels af te trekken
- Stap 18: Maak een binair gefilterde afbeelding
- Stap 19: Verwijder kleinere blobs gevonden in de gefilterde afbeeldingen
- Stap 20: Maak een schijfstructureringselement
- Stap 21: Voer morfologische afsluitingsbewerkingen uit
- Stap 22: Zoek de objecten met connectiviteit van ten minste 8
- Stap 23: Zoek het maximale aantal verbonden pixels
- Stap 24: Stel de maximale pixelwaarden in op 0 en zoek pixels met >=26 pixelconnectiviteit
- Stap 25: Bloedvaten in afbeelding verwijderen
- Stap 26: Figuurweergave
- Stap 27: Verwijder vaten en tel bloedvlekken
- Stap 28: Diagnose van het netvliesbeeld op basis van het aantal geïdentificeerde bloedstolsels
- Stap 29: Als er meer dan 5 blobs zijn…
- Stap 30: Herhaal het filterproces voor normale afbeeldingen met beeldcijferwaarden als 2 en 3
- Stap 31: Herhaal het hele proces voor de gediagnosticeerde afbeeldingen
- Stap 32: statistische analyse
- Stap 33: Betrouwbaarheidsinterval vinden
- Auteur John Day day@howwhatproduce.com.
- Public 2024-01-30 11:18.
- Laatst gewijzigd 2025-01-23 15:01.

(Zie codeoverzicht hierboven)
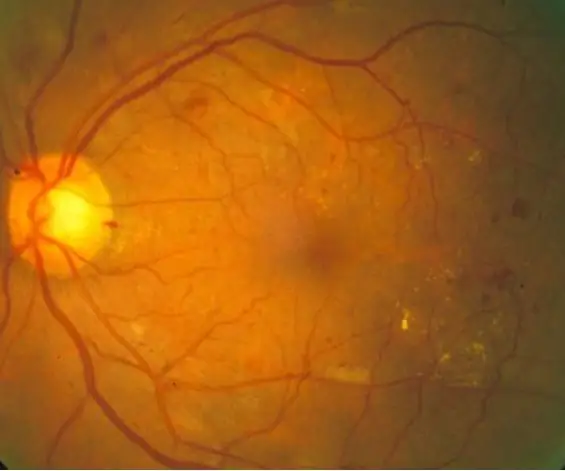
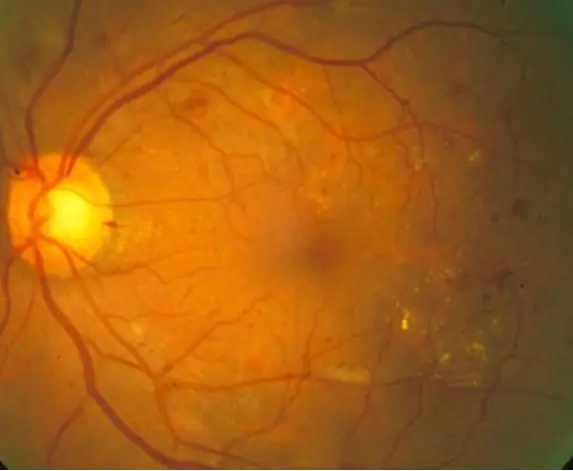
Diabetische retinopathie is een aan diabetes gerelateerde oogziekte die wordt veroorzaakt door een hoge bloedsuikerspiegel. De hoge bloedsuikerspiegels zorgen ervoor dat de bloedvaten in het netvlies opzwellen, wat leidt tot vergrote bloedvaten en zelfs bloedvatlekken, wat leidt tot donkere vlekken op retinale beelden. Met deze code willen we het verschijnen van bloedvatlekvlekken gebruiken als een indicator van diabetische retinopathie op de achtergrond, hoewel in de echte wereld verdere diagnosetechnieken vereist zouden zijn. Het doel van deze code is om beeldverwerking en diagnose van retinale beelden te automatiseren om tekenen van diabetische retinopathie te identificeren die worden weergegeven door donkere vlekken in de retinale beelden.
10 normale netvliesbeelden en 10 gediagnosticeerde netvliesbeelden werden verwerkt via een code die eerst de beelden leest en filtert en vervolgens de donkere vlekken kwantificeert om te bepalen of diabetische retinopathiesymptomen aanwezig zijn, op basis van een bepaalde drempel. De resultaten worden vervolgens afgedrukt op het opdrachtvenster voor interpretatie door de kijker.
Stap 1: Vereisten
1. Zorg ervoor dat u het MATLAB-programma op uw computer hebt gedownload.
2. Download het txt-bestand in de link. (Druk op 'ctrl+s' om op te slaan in dezelfde map als de MATLAB-code)
Stap 2: Vereisten (vervolg)
4. Open MATLAB en typ 'uiimport' in het opdrachtvenster.
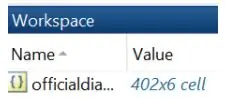
5. Selecteer het bestand officialdiagnoses.txt en importeer het in MATLAB als een celmatrix.
6. Zorg ervoor dat je ‘officiële diagnoses’ als variabele ziet in de werkruimte.
Stap 3: Vereisten (vervolg)

7. Download de functie ModWald.m, die bovenstaande code kan verkrijgen of download deze van Canvas.
(Code verstrekt door Professor King en Professor Choi)
Stap 4: Vereisten (vervolg)

8. Download de 400 onbewerkte afbeeldingen uit de datasectie van The STARE Project.
Stap 5: Matlab opruimen om u voor te bereiden op het uitvoeren van code

Toevoegen aan code:
1. sluit alles (sluit alle eerder geopende afbeeldingen)
2. clearvars - behalve officiële diagnoses (wist alle variabelen behalve het officiële diagnose-txt-bestand dat eerder is geïmporteerd)
3. cclc (wist opdrachtvenster)
Stap 6: Selecteer 10 normale oogbeelden en 10 beelden met diabetische retinopathiesymptomen
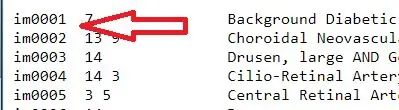
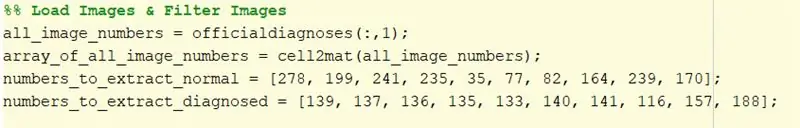
1. Pak het diagnosetekstbestand en pak de afbeeldingsnamen uit. Deze namen staan in de eerste kolom van het tekstbestand, dus typ 'officialdiagnoses(:, 1)' om ze uit te pakken. De matrix van afbeeldingsnamen is toegewezen aan een variabele, " all_image_numbers"
2. Converteer de variabele all_image_numbers van een celarray naar een matrixarray met behulp van de cell2mat-functie
Stap 7: Selecteer 10 normale oogbeelden en 10 beelden met diabetische retinopathiesymptomen (vervolg)
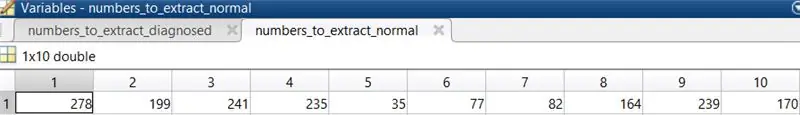
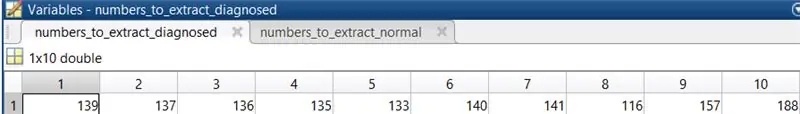
3. Selecteer 10 normale oogbeelden om de code uit te voeren. De geselecteerde afbeeldingen in dit geval waren 278, 199, 241, 235, 35, 77, 82, 164, 239, 170.
Plaats deze getallen in een matrix en wijs ze toe aan een variabele die wordt aangeroepen bij het laden van de afbeeldingen.
4. Herhaal stap 3 voor beelden van het netvlies waarbij diabetische retinopathie is vastgesteld. De geselecteerde afbeeldingen in dit geval waren 139, 137, 136, 135, 133, 140, 141, 116, 157, 188.
Stap 8: Maak 2 variabelen (normaal en gediagnosticeerd) en stel ze elk gelijk aan 0

Maak deze variabelen vóór de for-lus om de lusnummers te initialiseren.
Stap 9: Maak een for-lus om automatisch normale afbeeldingen te uploaden

1. Maak een for-lus
2. Stel een telvariabele (i, in dit geval) in op een matrix van waarden 1-10. Deze telvariabele wordt gebruikt om elke afbeelding afzonderlijk aan te roepen
3. Neem het i-element in de matrix van afbeeldingen om de afbeeldingsnaam te extraheren en om te zetten van een string naar een getal met behulp van de num2str-functie.
Zoek het aantal cijfers in de afbeeldingsnaam met behulp van de cijferfunctie. Wijs deze waarde toe aan een variabele, digits_normal. Dit nummer moet 1 zijn voor nummers met één cijfer, 2 voor nummers met twee cijfers en 3 voor nummers met drie cijfers. Deze informatie wordt gebruikt om automatisch afbeeldingen op te roepen.
Stap 10: Maak een for-lus om automatisch normale afbeeldingen te uploaden (vervolg)

3. Maak een if-statement met alle drie de mogelijkheden uit de vorige stappen. Als de afbeeldingsnaam 1 cijfer heeft, wordt de afbeelding "im000" genoemd, als deze 2 cijfers heeft, wordt de afbeelding "im00" genoemd en als deze er 3 heeft, wordt de afbeelding "im0" genoemd.
4. Wijs onder elk if-statement een variabele toe om "im" te imreaden onder het corresponderende if-statement met het juiste aantal nullen (zoals hierboven beschreven), gevolgd door de i.
Stap 11: Snijd de randen van de afbeelding bij
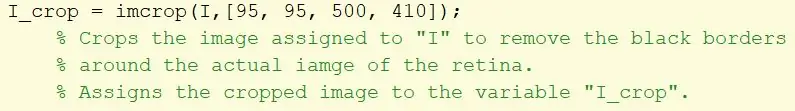
Neem de originele afbeelding en pas een imcrop-filter toe om zwarte randen te elimineren en wijs deze toe aan een variabele I_crop. De bijsnijdrechthoek wordt gespecificeerd met behulp van een matrix [95, 95, 500, 410].
Stap 12: Maak een grijsschaalafbeelding

Neem de bijgesneden afbeelding en pas het rbg2gray-filter toe om de afbeelding in grijswaarden te veranderen. Wijs deze afbeelding toe aan variabele I2.
Stap 13: Maak een contrasterende afbeelding
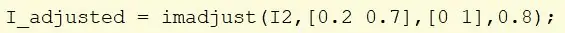
Neem de afbeelding I2 en gebruik imadjust om de intensiteitswaarden opnieuw te schalen.
Neem waarden die binnen het bereik [0,2, 0,7] vallen en schaal deze opnieuw naar een [0, 1]. Het gamma is ingesteld op 0,8 om het beeld helderder te maken. Wijs de nieuwe afbeelding toe aan I_adjusted.
Stap 14: Verbeter het contrastbeeld
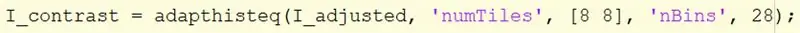
Neem de I_adjusted-afbeelding en gebruik de adapthisteq-functie om het contrast te verbeteren.
De Adapthisteq-syntaxis vereist de afbeeldingsnaam, I_adjusted, 'numTiles', de grootte van numTiles, 'nBins' en het aantal bins. De grootte van numTiles is ingesteld op [8 8], waarbij de afbeelding wordt verdeeld in tegels van 8x8 en het aantal bakken is ingesteld op 28. Wijs de afbeelding toe aan I_constrast.
Stap 15: Maak een gemiddeld filter

Maak een variabele met de naam 'meanfilt' met behulp van de functie fspecial. Voer 'gemiddelde functie' in om het middelingsfilter te maken en voer [90 90] in voor de schuifvenstergrootte.
Stap 16: Combineer het middelingsfilter met de contrasterende afbeelding
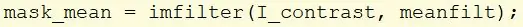
Maak een nieuwe variabele met de naam mask_mean en gebruik de functie imfilter om de afbeelding I_contrast te nemen en het eerder gemaakte gemiddelde filter toe te passen.
Stap 17: Maak een nieuw gemiddeld masker door pixels af te trekken

Maak een variabele met de naam mask_mean2 en gebruik de functie imsubtract om de waarde van elke pixel in I_contrast af te trekken van de overeenkomstige pixel in mask_mean.
Stap 18: Maak een binair gefilterde afbeelding

Zet grijswaardenafbeeldingen om in zwart-wit met behulp van imbinarize. Invoer mask_mean2, 'adaptive', 'ForegroundPolarity', 'dark', 'Sensitivity', 0.6. Wijs deze nieuwe afbeelding toe aan mask_binarize.
Stap 19: Verwijder kleinere blobs gevonden in de gefilterde afbeeldingen
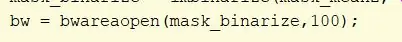
Verwijder objecten met een connectiviteit van minder dan 100 pixels met behulp van de bwareaopen-functie op de mask_binarize en stel de drempelwaarde in op 100. Wijs de variabele toe als bw.
Stap 20: Maak een schijfstructureringselement

Maak een schijfstructurerend element (met een straal van 2) met behulp van de strel-functie. Wijs het toe aan se.
Stap 21: Voer morfologische afsluitingsbewerkingen uit

Neem bw en pas de imclose-functie toe op het structurele element om een morfologische close-operatie op het object uit te voeren.
Stap 22: Zoek de objecten met connectiviteit van ten minste 8
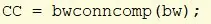
Neem bw en gebruik bwconncomp om de objecten te vinden met een connectiviteit van minimaal 8 in de afbeelding. Wijs de nummeruitvoer toe aan cc_1.
Stap 23: Zoek het maximale aantal verbonden pixels
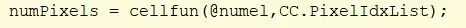
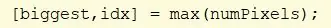
Gebruik de cellfun-functie om de functie "numel" op elke cel in CC uit te voeren. Hiermee wordt het aantal elementen in de PixelIdxList-cel gevonden. Waarde toewijzen aan "numPixels".
Zoek de maximale waarden in numPIxels. Wijs het grootste maximum toe aan "grootste" en de index van de maximale waarde aan "idx".
Stap 24: Stel de maximale pixelwaarden in op 0 en zoek pixels met >=26 pixelconnectiviteit
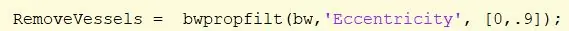



Zet de pixels met de grootste waarden in de afbeelding “bw” op 0, waardoor de pixels zwart worden.
Zoek de objecten met een connectiviteit van ten minste 26 pixels in de afbeelding met behulp van bwconncomp. Toewijzen aan variabele cc_1.
Stap 25: Bloedvaten in afbeelding verwijderen
Verwijder de bloedvaten die nog in het beeld aanwezig zijn met behulp van de bwpropfilt-functie met een bereik van [0, 0.9].
[0.9, 1] is uitgesloten omdat waarden dichtbij 1 een lijn aangeven. Wijs toe aan "VerwijderVessels".
Stap 26: Figuurweergave
Geef elke gefilterde afbeelding weer in een subplot. laat zien. met 'border' en 'strakke' ingangen, geeft elke afbeelding weer in een subplotstructuur. Voeg een titel toe aan elke afbeelding om te onderscheiden welk filter is gebruikt.
Stap 27: Verwijder vaten en tel bloedvlekken
1. Neem "RemoveVessels" en pas de functie 'Centroid' toe in regionprops om de zwaartepunten van de objecten in de afbeelding te identificeren. Deze objecten moeten overeenkomen met bloedstolsels die in het beeld aanwezig zijn.
2. Tel het aantal geïdentificeerde bloedstolsels door de lengte van de zwaartepuntmatrix te nemen.
Stap 28: Diagnose van het netvliesbeeld op basis van het aantal geïdentificeerde bloedstolsels
Gebruik if-statements om het beeld te diagnosticeren op basis van het aantal geïdentificeerde bloedstolsels.
Als het aantal geïdentificeerde zwaartepunten minder dan of gelijk aan 5 was, werd het beeld als normaal geïdentificeerd.
Als het aantal zwaartepunten groter was dan 5, werd het beeld gediagnosticeerd met diabetische retinopathie.
Het resultaat wordt met fprintf in het opdrachtvenster afgedrukt.
Stap 29: Als er meer dan 5 blobs zijn…
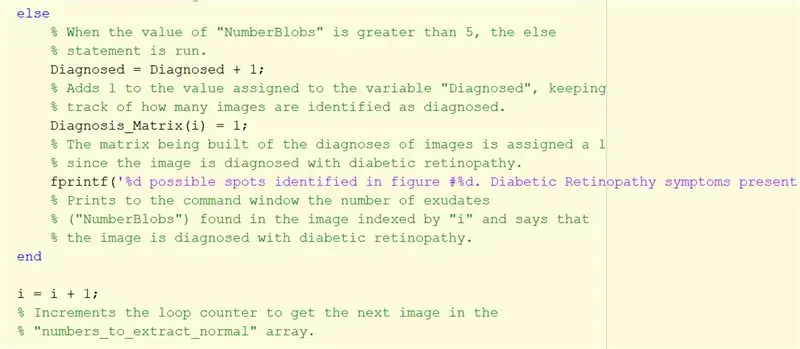
Herhaal de bovenstaande instructies voor gediagnosticeerde beelden als een else-statement. Dit deel wordt uitgevoerd als het aantal blobs groter is dan 5.
Beëindig het if-statement.
Stap 30: Herhaal het filterproces voor normale afbeeldingen met beeldcijferwaarden als 2 en 3
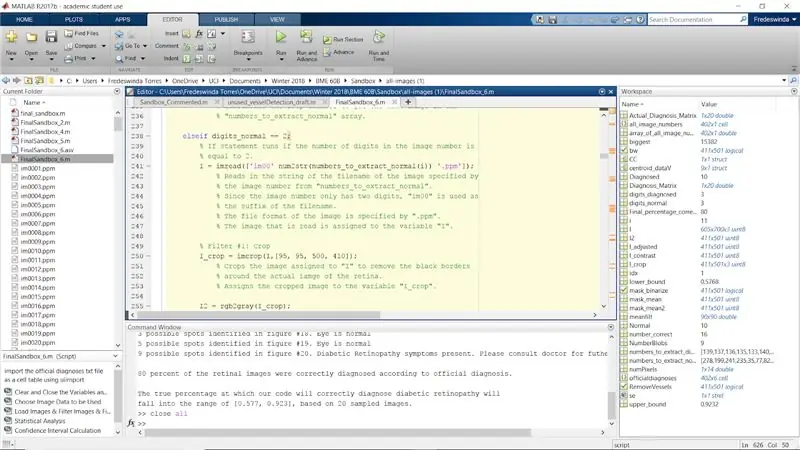
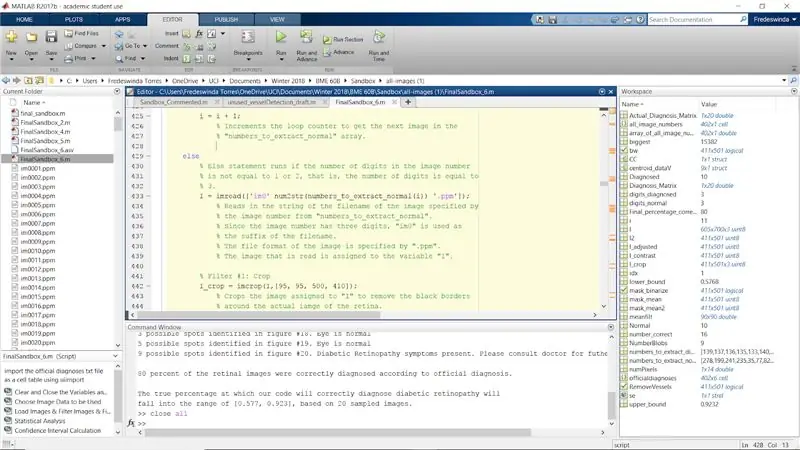
Herhaal het proces voor de rest van de originele if-statements wanneer numel (het aantal cijfers in het afbeeldingsnummer) gelijk is aan 2 en 3. Dit voltooit de for-lus voor de normale afbeeldingen.
Beëindig de for-lus.
Stap 31: Herhaal het hele proces voor de gediagnosticeerde afbeeldingen
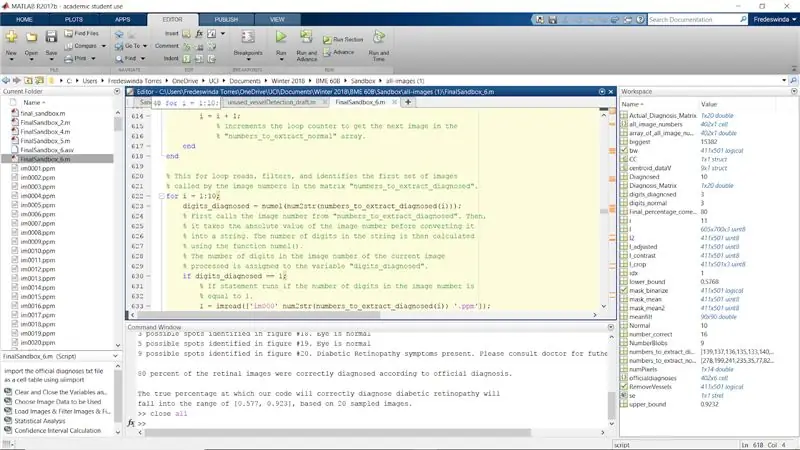
Herhaal het hele proces met behulp van de gediagnosticeerde beelden die worden vermeld in de matrix "numbers_to_extract_diagnosed".
Zorg ervoor dat u elk cijfer (i) doorloopt en verander het in cijfer (i+10), zodat de gediagnosticeerde cijfers verschijnen als afbeeldingen 11 tot 20.
Stap 32: statistische analyse
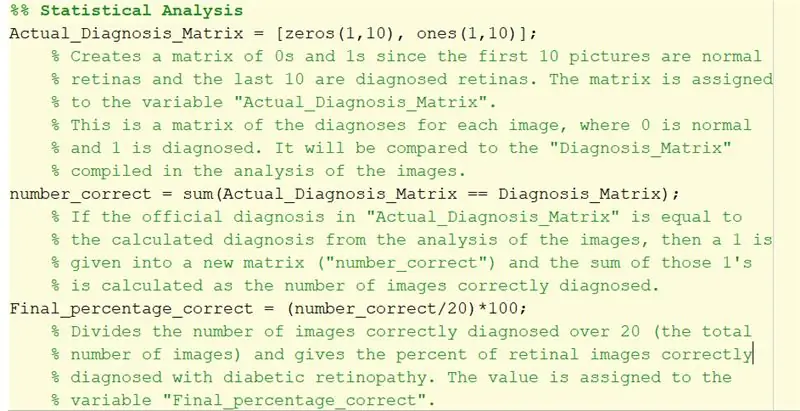
1. 'Actual_Diagnosis_Matrix' wordt gebruikt om de resultaten te vergelijken met de officiële diagnose in het txt-bestand. De eerste 10 nullen geven aan dat de eerste 10 afbeeldingen normaal moeten zijn. De laatste 10 geven aan dat de laatste 10 beelden moeten worden geclassificeerd als diabetische retinopathie.
2. Het dubbele gelijkteken dat wordt gebruikt om 'number_correct' te creëren, creëert een logische array door de waarde van de corresponderende elementen van 'Actual_Diagnosis_Matrix' te vergelijken met 'Diagnosis_Matrix' gemaakt op basis van de for-lus.
Voor elk element dat overeenkomt met de diagnose wordt een 1 toegevoegd, wat betekent dat de code die afbeelding correct diagnosticeert. Als het onjuist is, wordt een 0 aan de matrix toegevoegd.
Als je de som daarvan neemt, worden alle getallen bij elkaar opgeteld. Met andere woorden, het vindt de som van correct gediagnosticeerde beelden.
3. 'Final_percentage_correct' is het berekende percentage van hoe nauwkeurig de code diabetische retinopathie diagnosticeerde. Het aantal correct gediagnosticeerde beelden wordt gedeeld door 20 (het totale aantal foto's) en vermenigvuldigd met 100 om het percentage succesvolle diagnoses te vinden.
Stap 33: Betrouwbaarheidsinterval vinden
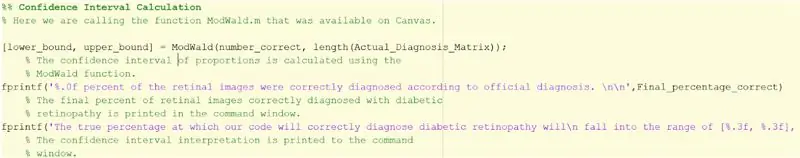
1. Zorg ervoor dat je ModWald.m hebt gedownload om het als een functie aan te roepen. Zonder de functie zou u het betrouwbaarheidsinterval zelf moeten berekenen met de aangepaste Wald-methode.
2. De ModWald-functie heeft 2 ingangen waarbij de eerste het aantal correct geïdentificeerde afbeeldingen is en de tweede het totale aantal afbeeldingen.
3. De ModWald-functie geeft de onder- en bovengrenzen van het betrouwbaarheidsinterval van verhoudingen weer voor de nauwkeurigheid van de gesamplede gegevens. Met andere woorden, het geeft u een interval van percentages waar het echte percentage van de nauwkeurigheid van de code zal liggen.
4. Gebruik onderstaande fprintf om de statistieken en het betrouwbaarheidsinterval uit te voeren in het opdrachtvenster.
> fprintf('%.0f procent van de retinale beelden werd correct gediagnosticeerd volgens de officiële diagnose. \n\n', Final_percentage_correct)
> fprintf('Het werkelijke percentage waarmee onze code diabetische retinopathie correct zal diagnosticeren, zal\n binnen het bereik van [%.3f, %.3f] vallen, gebaseerd op 20 gesamplede afbeeldingen \n', lower_bound, upper_bound)
Aanbevolen:
Harde schijven: diagnose, probleemoplossing en onderhoud: 3 stappen

Harde schijven: diagnose, probleemoplossing en onderhoud: wat is een harde schijf? - Simpel gezegd, op de harde schijf worden al uw gegevens opgeslagen. Het herbergt de harde schijf, waar al uw bestanden en mappen zich fysiek bevinden. De informatie wordt magnetisch op de schijf opgeslagen, zodat deze op de schijf blijft, zelfs wanneer
BMW INPA E60 Codering/Diagnose Gids: 4 Stappen

BMW INPA E60 Coderings- / Diagnosegids: In dit artikel zal ik enkele gevallen verzamelen over de BMW INPA-werkkoffer op BMW E60, en als u een andere BMW INPA E60-zaak met mensen wilt delen, stuur dan naar mijn e-mail.I zal het hier plaatsen om het te delen
Apparaten bedienen via internet via browser. (IoT): 6 stappen

Apparaten bedienen via internet via browser. (IoT): In deze Instructable zal ik je laten zien hoe je apparaten zoals LED's, relais, motoren etc. via internet via een webbrowser kunt bedienen. En u hebt via elk apparaat veilig toegang tot de bedieningselementen. Het webplatform dat ik hier heb gebruikt, is RemoteMe.org bezoek
Instructies voor het voltooien van de mock-up van het ontwerp van de rupsschuif voor het heffen/verlagen van in het midden gemonteerde voetsteunen op elektrische rolstoelen - Ajarn

Instructies voor het voltooien van de mock-up van het ontwerp van de rupsschuif voor het omhoog/omlaag brengen van in het midden gemonteerde voetsteunen op elektrische rolstoelen: in het midden gemonteerde voetsteunen kunnen omhoog worden gebracht om onder de zitting goed te worden opgeborgen en omlaag om te worden ingezet. Een mechanisme voor onafhankelijke bediening van het opbergen en plaatsen van de voetsteun is niet inbegrepen in elektrische rolstoelen op de markt, en PWC-gebruikers hebben de behoefte geuit
Inventaris - Snelle pc-diagnose nu vervangen, zie hieronder: 6 stappen

Inventaris - Snelle pc-diagnose nu vervangen, zie hieronder: Als u ooit op een pc moet werken die niet voor u ligt, heeft u een nauwkeurige inventaris nodig. Deze instructable is puur ontstaan omdat een collega moeite heeft met het vinden van een driver voor een pc. Ik zal Aida32 persoonlijk gebruiken van http://majorgeeks.com
