
Inhoudsopgave:
- Auteur John Day [email protected].
- Public 2024-01-30 11:16.
- Laatst gewijzigd 2025-01-23 15:01.

ThinkBioT is ontworpen om "Plug and Play" te zijn, met Edge TPU-compatibele TensorFlow Lite-modellen.
In deze documentatie behandelen we het maken van spectrogrammen, het formatteren van uw gegevens en het gebruik van Google AutoML.
De code in deze tutorial wordt in bash geschreven en is dus compatibel met meerdere platforms.
afhankelijkheden
- Voordat u begint, moet u Sox echter een opdrachtregelaudioprogramma installeren dat compatibel is met Windows-, Mac- en Linux-apparaten.
- Als je een Windows-apparaat gebruikt, is de gemakkelijkste manier om bash-scripts uit te voeren via Git, dus ik zou aanraden en downloaden en installeren omdat het op veel manieren nuttig is,
- Gebruik voor het bewerken van code uw favoriete editor of installeer NotePad++ voor Windows of Atom voor andere besturingssystemen.
**Als je een bestaand TensorFlow-model hebt of je wilt leren overdragen met een bestaand model, raadpleeg dan de Google Coral-documentatie.
Stap 1: Stel een Google Cloud Storage-bucket in
1. Log in met je gmail-account (of maak er een aan als je geen Google-account hebt)
2. Ga naar de projectselectiepagina en maak een nieuw project voor je model- en spectrogrambestanden. U moet facturering inschakelen om verder te kunnen gaan.
3. Ga naar https://cloud.google.com/storage/ en druk op de knop Create bucket bovenaan de pagina.
4. Voer de gewenste bucketnaam in en maak de bucket die de standaardinstellingen accepteert.
Stap 2: Formatteer uw gegevens en maak een dataset Csv
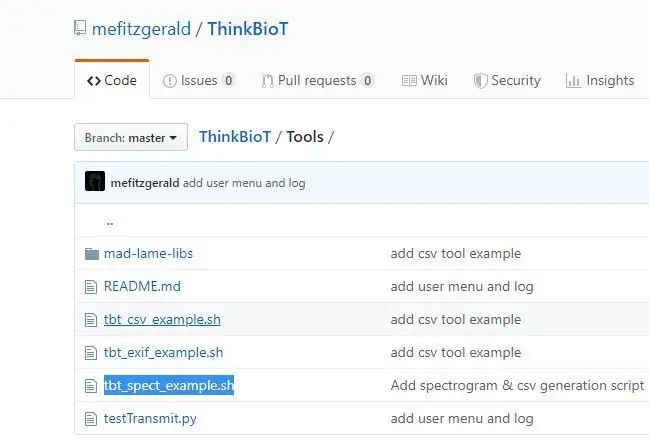
Ik heb een handig script ontworpen om uw dataset.csv-bestand te maken dat nodig is om uw model te maken. Het datasetbestand koppelt de afbeeldingen in uw bucket aan hun labels in de dataset.
1. Download de ThinkBioT-repository van GitHub en
2. Kopieer het bestand tbt_spect_example.sh van de map Tools naar een nieuwe map op uw bureaublad.
3. Voeg de audiobestanden toe die u in uw model wilt gebruiken en plaats ze in mappen met hun label (dwz waarin u ze wilt sorteren. Als u bijvoorbeeld honden of katten wilt identificeren, kunt u een map hebben hond, met blafgeluiden OF map met de naam kat met kattengeluiden enz.
4. Open de tbt_spect_example.sh met Notepad++ en vervang "yourbucknamename" in regel 54 door de naam van uw Google Storage Bucket. Als uw bucket bijvoorbeeld myModelBucket heette, zou de regel worden gewijzigd in:
bucket="gs://myModelBucket/spectro-data/"
5. Voer de code uit door het volgende in uw Bash-terminal te typen, de code wordt uitgevoerd en maakt uw labels csv-bestand en een map met de naam spectro-data op uw bureaublad met de resulterende spectrogrammen.
sh tbt_spect_example.sh
Stap 3: Upload uw spectrogrammen naar uw bucket
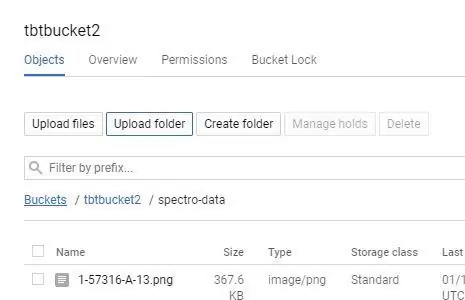
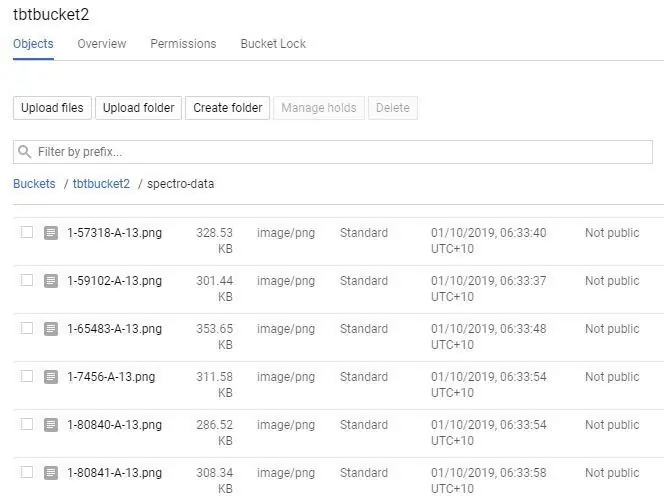
Er zijn een paar manieren om te uploaden naar Google Storage, de gemakkelijkste is om een directe map te uploaden;
1. Klik op uw bucketnaam op uw Google Storage-pagina.
2. Selecteer de "UPLOAD FOLDER"-knop en kies uw "spectro-data/"-directory die u in de laatste stap hebt gemaakt.
OF
2. Als u een grote hoeveelheid bestanden heeft, kunt u handmatig de map "spectro-data/" maken door "MAP MAKEN" te selecteren, vervolgens naar de map navigeren en "BESTANDEN UPLOADEN" selecteren. Dit kan een geweldige optie zijn voor grote datasets, omdat je de spectrogrammen in secties kunt uploaden, zelfs als je meerdere computers gebruikt om de uploadsnelheid te verhogen.
OF
2. Als je een gevorderde gebruiker bent kun je ook uploaden via Google Cloud Shell;
gsutil cp spectro-data/* gs://uw-bucket-naam/spectro-data/
Je zou nu een emmer vol met mooie spectrogrammen moeten hebben!
Stap 4: Upload uw dataset Csv
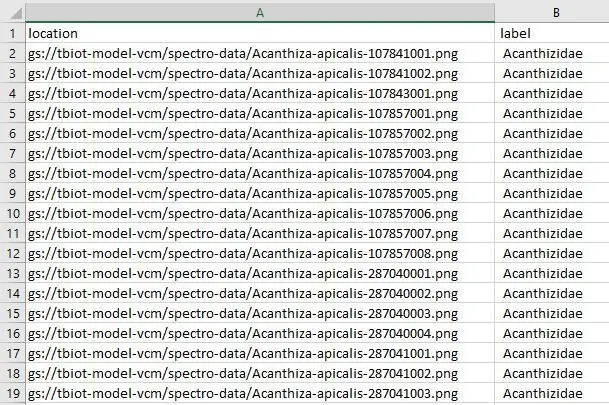
Nu moeten we het model-labels.csv-bestand uploaden naar uw "spectro-data/"-directory in Google Storage, het is in wezen hetzelfde als de laatste stap, u uploadt slechts een enkel bestand in plaats van veel.
1. Klik op uw bucketnaam op uw Google Storage-pagina.
2. Selecteer de UPLOAD FILE-knop en kies uw model-labels.csv-bestand dat u eerder hebt gemaakt.
Stap 5: Gegevensset maken
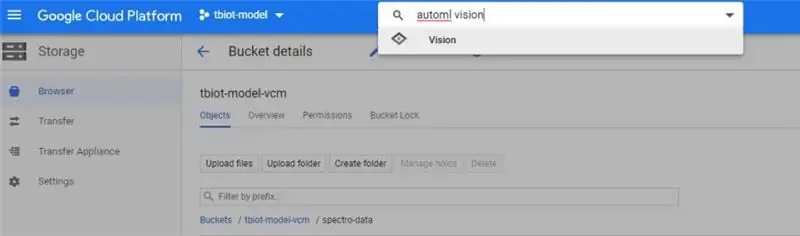
1. Eerst moet u de AutoML VIsion API vinden, dit kan een beetje lastig zijn! De eenvoudigste manier is om te zoeken naar "automl vision" in de zoekbalk van uw Google Cloud-opslag (afgebeeld).
2. Zodra u op de API-link klikt, moet u de API inschakelen.
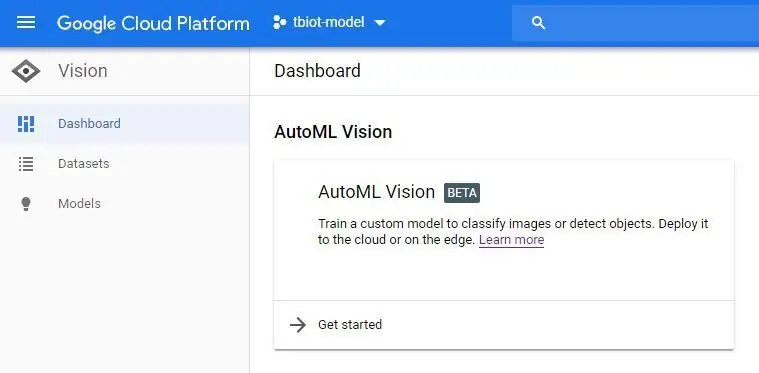
3. U bevindt zich nu in het AutoML Vision Dashboard (afgebeeld) klik op de nieuwe dataset-knop en selecteer Enkel label en de optie 'Selecteer een CSV-bestand'. U neemt dan de link naar uw model-labels.csv-bestand op in uw opslagbucket. Als je deze tutorial hebt gevolgd, is het zoals hieronder:
gs://yourBucketName/spectro-data/model-labelsBal.csv
4. Druk vervolgens op doorgaan om uw dataset aan te maken. Het maken kan even duren.
Stap 6: Maak uw AutoML-model
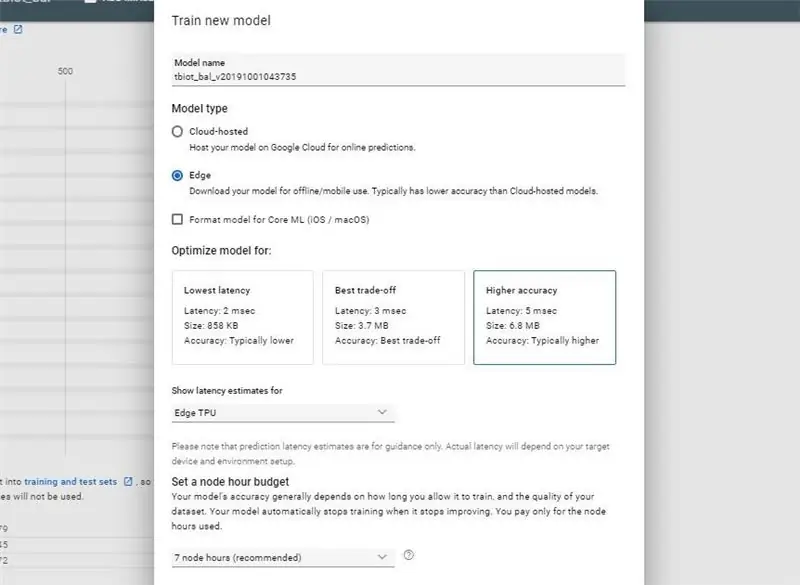

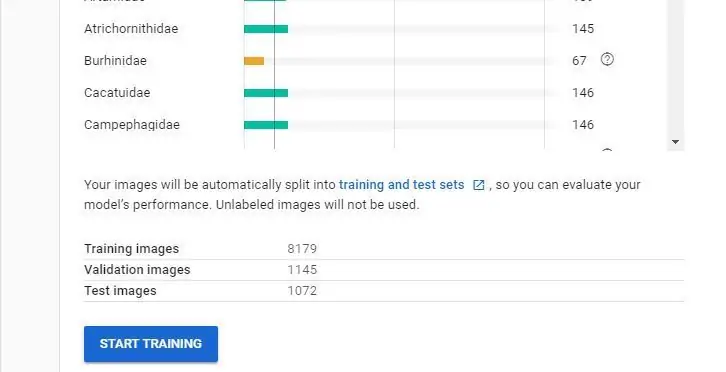
Zodra u uw e-mail heeft ontvangen om u te laten weten dat uw dataset is gemaakt, bent u klaar om uw nieuwe model te maken.
- Druk op de TRAIN-knop
- Selecteer modeltype: Edge en Model latentie schattingen: Edge TPU en laat de andere opties in eerste instantie als standaard, misschien wil je er later mee experimenteren.
- Nu gaat je model trainen, het duurt even en je ontvangt een e-mail wanneer het klaar is om te downloaden.
Opmerking: als je trainingsknop niet beschikbaar is, heb je mogelijk problemen met je dataset. Als je minder dan 10 van elke klasse (label) hebt, laat het systeem je geen model trainen, dus het kan zijn dat je extra afbeeldingen moet toevoegen. Het is de moeite waard om de Google AutoML-video te bekijken als u opheldering nodig heeft.
Stap 7: Test uw model
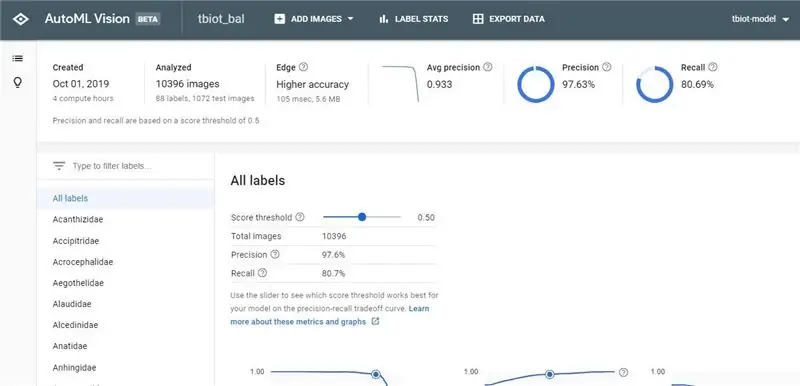
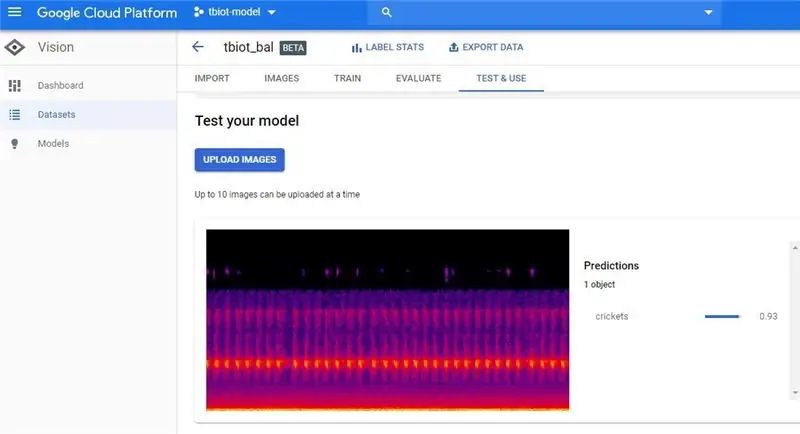
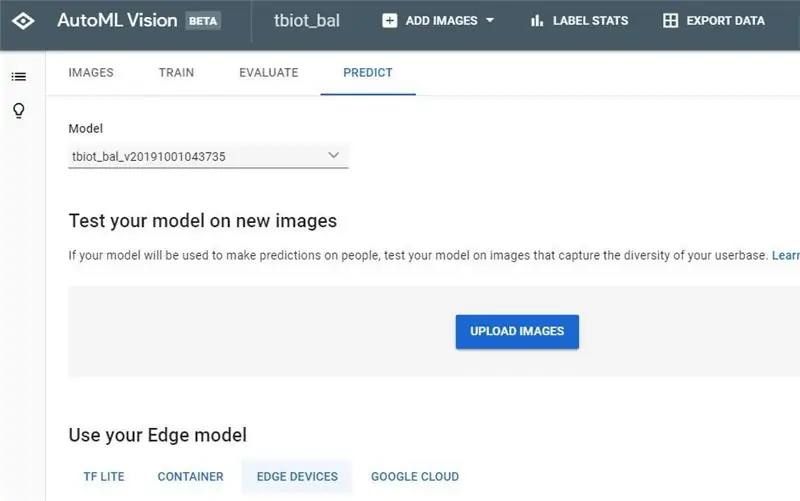
Zodra u uw e-mail voor het voltooien van uw model heeft ontvangen, klikt u op de link om terug te keren naar de AutoML Vision API.
1. Nu kunt u uw resultaten en de verwarringsmatrix voor uw model bekijken.
2. De volgende stap is om je model te testen, ga naar 'TEST & GEBRUIK' of 'PREDICT'. Vreemd genoeg lijken er 2 gebruikers-GUI's te zijn, die ik beide heb afgebeeld, maar de opties hebben beide dezelfde functionaliteit.
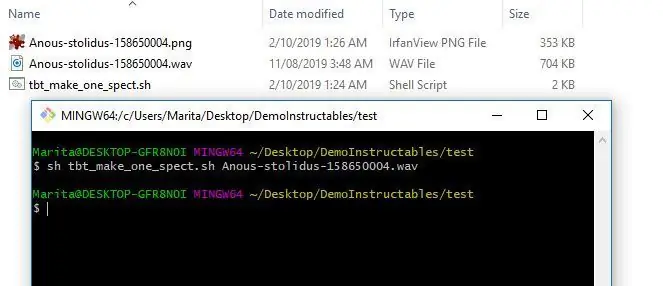
3. Nu kunt u een testspectrogram uploaden. Om een enkel spectrogram te maken, kun je het programma tbt_make_one_spect.sh van de ThinkBioT Github gebruiken. Zet het gewoon in een map met de wav die je naar een spectrogram wilt converteren, open een Git Bash-venster (of terminal) en gebruik de onderstaande code, ter vervanging van je bestandsnaam.
sh tbt_make_one_spect.sh yourWavName.wav
4. Upload nu eenvoudig het spectrogram en controleer uw resultaat!
Stap 8: installeer uw model in ThinkBioT
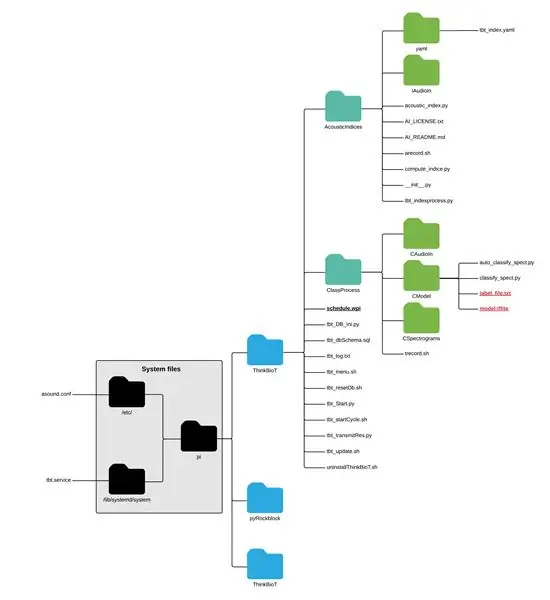
Om uw nieuwe glanzende model te gebruiken, laat u het model en het txt-bestand gewoon in de CModel-map vallen;
pi > ThinkBioT > ClassProcess > CModel
Nu ben je klaar om ThinkBioT te gebruiken:)
**NB** Als u uw model buiten het ThinkBioT-framework gebruikt, moet u uw labeldocument bewerken door umbers toe te voegen aan het begin van elke regel, aangezien de nieuwste tflite-interpreters ingebouwde "readlabels"-functie ervan uitgaat dat ze er zijn. Ik heb een aangepaste functie geschreven in het ThinkBioT-framework classify_spect.py als een work around die je in je eigen code mag gebruiken:)
def ReadLabelFile(file_path):
counter = 0 met open(file_path, 'r', encoding='utf-8') als f: lines = f.readlines() ret = {} voor regel in regels: ret[int(counter)] = line.strip () teller = teller + 1 retour ret
Aanbevolen:
Door beweging geactiveerde cosplayvleugels met Circuit Playground Express - Deel 1: 7 stappen (met afbeeldingen)

Door beweging geactiveerde cosplayvleugels met Circuit Playground Express - Deel 1: Dit is deel één van een tweedelige project, waarin ik je mijn proces laat zien voor het maken van een paar geautomatiseerde feeënvleugels. Het eerste deel van het project is de mechanica van de vleugels, en het tweede deel is het draagbaar maken en de vleugels toevoegen
Deel 1. ThinkBioT Autonomous Bio-akoestische Sensor Hardware Build - Ajarnpa

Deel 1. ThinkBioT Autonomous Bio-acoustic Sensor Hardware Build: ThinkBioT heeft tot doel een software- en hardwareraamwerk te bieden, ontworpen als een technologische ruggengraat om verder onderzoek te ondersteunen, door de details van gegevensverzameling, voorverwerking, gegevensoverdracht en visualisatietaken uit te voeren die onderzoeker
Hoe u uw eerste actie voor Google Home kunt bouwen (in 10 minuten) Deel-1: 10 stappen
Hoe u uw eerste actie voor Google Home kunt bouwen (in 10 minuten) Deel 1: Hallo, dit is de eerste in een reeks artikelen die ik ga schrijven waarin we zullen leren hoe we acties op Google kunnen ontwikkelen en implementeren. Eigenlijk ben ik bezig met “acties op google” van de afgelopen maanden. Ik heb veel artikelen doorgenomen die beschikbaar zijn op
Hoe u uw eigen windmeter kunt bouwen met behulp van reed-schakelaars, Hall-effectsensor en wat restjes op Nodemcu. - Deel 1 - Hardware: 8 stappen (met afbeeldingen)

Hoe u uw eigen windmeter kunt bouwen met behulp van reed-schakelaars, Hall-effectsensor en wat restjes op Nodemcu. - Deel 1 - Hardware: Inleiding Sinds ik ben begonnen met de studies van Arduino en de Maker Culture, heb ik het leuk gevonden om nuttige apparaten te bouwen met behulp van rommel en afvalstukken zoals kroonkurken, stukjes PVC, drankblikjes, enz. Ik geef graag een tweede leven aan elk stuk of elke partner
Draadloze communicatie met behulp van goedkope 433MHz RF-modules en Pic-microcontrollers. Deel 2: 4 stappen (met afbeeldingen)

Draadloze communicatie met behulp van goedkope 433MHz RF-modules en Pic-microcontrollers. Deel 2: In het eerste deel van deze instructable demonstreerde ik hoe je een PIC12F1822 programmeert met behulp van MPLAB IDE en XC8-compiler, om een eenvoudige string draadloos te verzenden met goedkope TX/RX 433MHz-modules. De ontvangermodule was via een USB aangesloten op UART TTL kabel advertentie
