Inhoudsopgave:
- Stap 1: Brain Box gebruiken
- Stap 2: Intro: dwarsdoorsneden analyseren
- Stap 3: Een functie instellen: LevelCurveTracings.m
- Stap 4: Het grensalgoritme ontwikkelen: grenzen vinden
- Stap 5: Het Bound Algoritme ontwikkelen: Outer Bound Array genereren
- Stap 6: Ontwikkelen van het gebonden algoritme: werken met middelpunt
- Stap 7: Ontwikkelen van het gebonden algoritme: wanneer een zwaartepunt niet gecentreerd is
- Stap 8: Ontwikkelen van het gebonden algoritme: interferentie van gaten
- Stap 9: Het Bound Algoritme ontwikkelen: Gaten lokaliseren, Brain Bounds en Hole Bounds afronden
- Stap 10: Gegevens loggen: Functie PatientFiles.m
- Stap 11: Gegevens in bestanden loggen
- Stap 12: Gegevens loggen: een plot van hersenvolume weergeven in de loop van de tijd
- Stap 13: hiaten in subplots dichten: Subplotclose.m
- Stap 14: De hoofdcode: alles wissen en vragen om invoer
- Stap 15: De hoofdcode: batchverwerking van de afbeeldingen
- Stap 16: De hoofdcode: opvulling
- Stap 17: De hoofdcode: grenzen bepalen
- Stap 18: De hoofdcode: de juiste Z-dimensie bepalen
- Stap 19: De hoofdcode: X- en Y-coördinaten bepalen
- Stap 20: De hoofdcode: een driedimensionale structuur uitzetten, volume vinden en gegevens loggen
- Auteur John Day day@howwhatproduce.com.
- Public 2024-01-31 10:23.
- Laatst gewijzigd 2025-01-23 15:01.
De opmars naar de grens van een langer menselijk leven heeft geleid tot de opkomst van ziekten die niet door beschavingen vóór de onze werden gezien. Hiervan trof de ziekte van Alzheimer in 2017 ongeveer 5,3 miljoen levende oudere Amerikanen, of ongeveer 1 op de 10 oudere Amerikanen (https://www.alz.org/facts/) en talloze anderen met dementie. Om te helpen bij de strijd om te begrijpen wat onze ouderen treft, zal deze code de toekomstige onderzoekers en nieuwsgierige nieuwsgierigheid uitrusten met de mogelijkheid om het hersenvolume in de loop van de tijd te volgen.
Stap 1: Brain Box gebruiken
Om Brain Box te gebruiken, heb je alleen het volgende nodig:
- MRI-scans van een brein en de naam en het formaat van dergelijke bestanden (moeten allemaal ongeveer dezelfde afmetingen hebben)
- Lengte van één scan
- Afstand tussen elke laag (MRI-scan)
- Patiëntnaam (Neem bij het invoeren geen spaties op en schrijf de voornaam en achternaam in hoofdletters, bijvoorbeeld: VoornaamAchternaam)
En op basis hiervan heeft men de mogelijkheid om individuele trends in hersenvolume in de loop van de tijd bij te houden. Zo kunnen met deze software cijfers over de trends van Alzheimer worden gevolgd. De lengte die we in de proef gebruikten was 180 mm voor de lengte van één scan en 5 mm voor de afstand tussen MRI-scans, gebaseerd op gemiddelde cijfers.
De toepassing van brainbox hoeft echter niet beperkt te blijven tot deze ene taak. Als de dwarsdoorsneden van een bepaalde vaste stof foto zijn, zoals een tumor op zich, kunnen de trends in volumeveranderingen voor deze ook in de software worden gevolgd.
Stap 2: Intro: dwarsdoorsneden analyseren
In driedimensionale structuren zouden de tweedimensionale vlakken waaruit deze bestaan, dwarsdoorsneden worden genoemd. Stel je voor dat een stapel papieren een rechthoekig prisma vormt, dan zou elk stuk papier een dwarsdoorsnede van het papier zijn. Bij het verbeelden van de hersenen passen we dezelfde gedachtegang toe. MRI (magnetic resonance imaging) (zie informatie over MRI) legt de dwarsdoorsneden van de hersenen vast en door de grenzen te gebruiken die zijn gedefinieerd in elke "laag" van de verstrekte hersenen, kunnen we een structuur construeren om het volume van de hersenen te modelleren en te vinden. We moeten echter eerst een functie bouwen om informatie over dergelijke grenzen te geven.
Stap 3: Een functie instellen: LevelCurveTracings.m
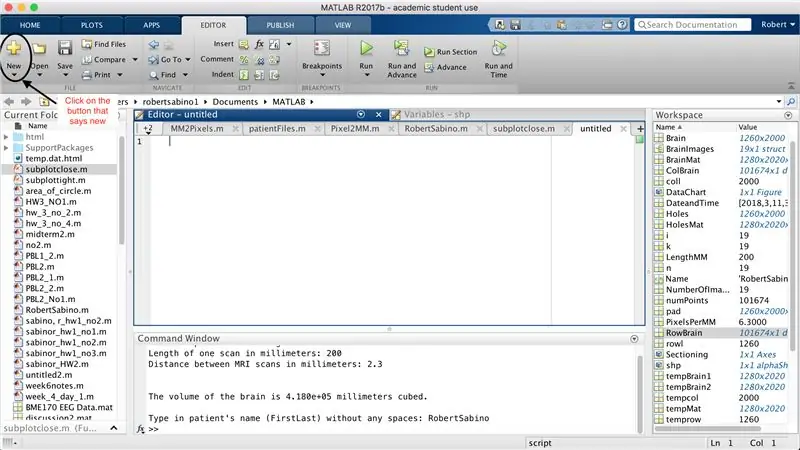
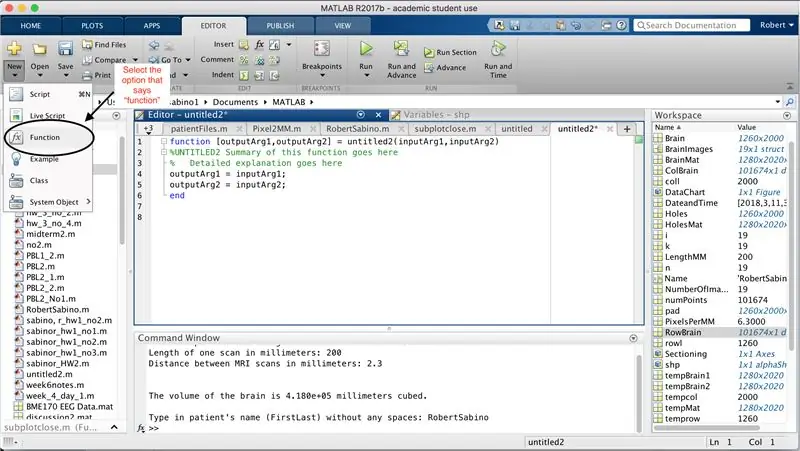
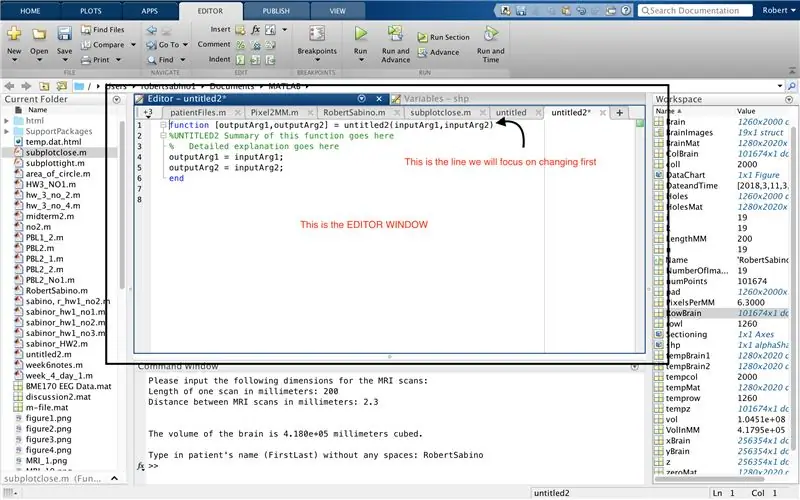

Zorg er eerst voor dat uw computer MATLAB_R2017b heeft gedownload (download hier) en open MATLAB. Klik in de MATLAB-interface op de knop in de linkerbovenhoek van het venster met de tekst "Nieuw" met een vet geel plusteken en selecteer de optie "functie", om in het editorvenster een ruimte te openen die lijkt op die in de derde foto. We zullen ons concentreren op het wijzigen van de eerste regel om de functie in te stellen. Waar staat "outputArg1", vervang het door "brain", "outputArg2" om "holes", "untitled2" voor "exp2" en "inputArg1" voor "image" te zeggen en verwijder "inputArg2". Je hebt nu een functie die moet worden aangeroepen met behulp van "exp2", waarbij je één argument "image" neemt en de grenzen van "brain" en "holes" uitvoert. De eerste regel van de functie moet lijken op de regel die wordt weergegeven in de vierde afbeelding. Verwijder alle code onder deze eerste regel.
Stap 4: Het grensalgoritme ontwikkelen: grenzen vinden

Typ de code als volgt onder de regel. Dit gedeelte van de functie doet het volgende regel voor regel.
- Laad in de afbeelding "image" in de variabele "mri".
- Verander "mri" in een afbeelding gemaakt van waarden in een reeks van getallen tot enen en nullen (ook wel binair genoemd) op basis van een ingestelde drempelwaarde. Als de waarde in een pixel gelijk is aan of groter is dan 0,1, wordt deze ingesteld op één, zo niet, dan wordt de waarde op die pixel op nul gezet.
- De volgende vier regels veranderen 10 kolommen en rijen aan de randen van de MRI-scanlaag in nullen, om te voorkomen dat onjuiste waarden worden gelezen als grenzen (zoals geleerd door experimenteren met de code).
- In de laatste regel traceert bwboundaries de grenzen van de binaire afbeelding "mri" en stelt deze gelijk aan "b", een array met de elementen waarvan de indices overeenkomen met die van de grenzen die zijn ingesteld op één.
Stap 5: Het Bound Algoritme ontwikkelen: Outer Bound Array genereren
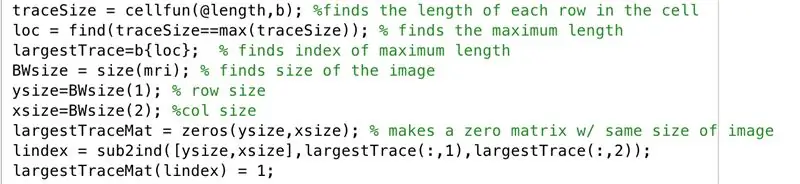
Volg mee in het editorvenster met de volgende code in de afbeelding. Dit gedeelte van de code doet het volgende regel voor regel.
- Zoek de lengte van elk van de rijen van de binaire afbeelding "b" (cellfun past de functielengte toe op elke rij).
- Stel "loc" in om de maximale lengtes op te slaan.
- Zoek de index van de maximale lengte, ingesteld om op te slaan in "largestTrace".
- Zoek de grootte van de afbeelding "mri", die uit dezelfde grootte bestaat als "b", en stel in op "BWsize".
- Zoek het aantal rijen in de array van de afbeelding, ingesteld op "ysize".
- Zoek het aantal kolommen in de array van de afbeelding, ingesteld op "xsize".
- Genereer array "largestTraceMat", een "ysize" door "xsize" matrix van nullen.
- Zoek de equivalente index van de geabonneerde waarden die overeenkomen met waar de grootste Trace x- en y-waarden waren, sla op in vector "lindex".
- Verander in de matrix van nullen, "largestTraceMat", de elementen bij de indices die overeenkomen met indexwaarden die zijn opgeslagen als elementen in "lindex" in enen.
Dus de logische array "largestTraceMat" heeft het grootste begrensde gebied van de gegeven hersenscandwarsdoorsnede uitgezet als enen met een achtergrond van nullen
Stap 6: Ontwikkelen van het gebonden algoritme: werken met middelpunt

Vervolgens moeten we testen of de dwarsdoorsnede uit meer dan één regio (de grootste) bestaat. Door de uitlijning van het zwaartepunt van de grootste regio te testen, kunnen we zien of er één aangrenzend gebied is, wat een meer gecentreerd zwaartepunt zou opleveren, of de mogelijkheid van meerdere regio's.
- Gebruik "regionProps" om informatie te vinden over de aanwezige zwaartepunten, stel gelijk aan de structuurarray "tempStruct"
- Vorm array "zwaartepunten" met gegevens uit het veld "zwaartepunt" verticaal aaneengeschakeld
- Neem de waarden in de tweede kolom van "zwaartepunten" (de coördinaten van de horizontale dimensie)
- Voer een filter uit om de uitlijning van het zwaartepunt ten opzichte van het horizontale midden te controleren
Stap 7: Ontwikkelen van het gebonden algoritme: wanneer een zwaartepunt niet gecentreerd is
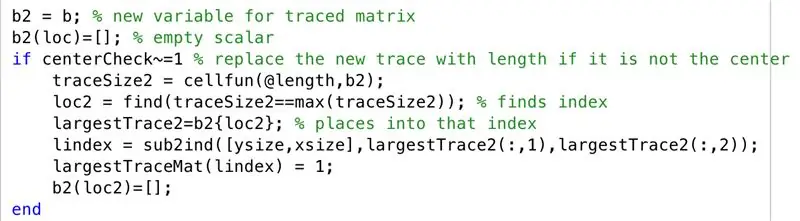
In het scenario dat het zwaartepunt van het grootste spoorgebied niet gecentreerd is, doorlopen we de volgende stappen. Zoals we in de MRI-scans hadden waargenomen, bestond de neiging om hersenhelften in de dwarsdoorsnede af te beelden als ze niet aaneengesloten waren, dus gaan we nu verder met het plotten van het op één na grootste spoor samen met het grootste spoor in "grootsteTraceMat"
- Stel de getraceerde matrix in op een nieuwe variabele "b2"
- Initialiseer lege matrix "b2", met een set geïndexeerd door "loc"
- Maak een voorwaarde, voor wanneer een zwaartepunt niet gecentreerd is (d.w.z. een laag met meerdere regio's)
- Stel voor elke rij een nieuwe traceergrootte in (traceSize2)
- Stel "loc2" in om de indices te vinden waar grenzen aanwezig zijn
- Laat cellen gespecificeerd door "loc2" in "b2" gelijk zijn aan "largestTrace2"
- Converteer de subscripts naar indices, ingesteld op "lindex"
- Wijzig elementen die overeenkomen met "lindex" in "largestTraceMat" naar 1
- Initialiseer lege matrix "b2", met een set geïndexeerd door "loc2"
Stap 8: Ontwikkelen van het gebonden algoritme: interferentie van gaten

Bij het omgaan met gaten, hielden de waarden die zijn opgeslagen in "b2" andere structuren bij dan het grootste spoor, en door deze uit te zetten op een gevulde vorm van "grootste TraceMat" zal onthullen waar er gaten in de hersengebieden zijn.
- Maak een array "filledMat", een ingevulde vorm van "largestTraceMat"
- Maak array "interferenceMat", een "ysize" bij "xsize" array van nullen
- Maak een array "interferenceloc", om de waarden van "b2" op te slaan, verticaal aaneengeschakeld
- Maak array "lindex" om de indices op te slaan die overeenkomen met "interferenceloc"
- Voor indices in "interferenceMat" die overeenkomen met "lindex", stelt u de waarde in op 1, waardoor een ander begrensd gebied wordt gemaakt
Stap 9: Het Bound Algoritme ontwikkelen: Gaten lokaliseren, Brain Bounds en Hole Bounds afronden

- Stel array "tempMat" in op "interferenceMat" plus "filledMat", waardoor elke waarde in de matrix bij elkaar wordt opgeteld
- Stel array "holesLoc" gelijk in aan de indices waarbij "interferenceMat" en "filledMat" beide gelijk waren aan één
- Stel "holesMat" in als een nulmatrix van afmetingen "ysize" x "xsize"
- Stel indices in "holesMat" in die gelijk zijn aan "holesLoc" als enen
- Zet "brain" op "largestTraceMat"
- Zet "gaten" op "gatenMat"
Met de bevinding waarbij de waarden van de toegevoegde matrices gelijk waren aan 2, konden de gatenlocaties gemakkelijk worden vastgezet en op een lege matrix worden uitgezet.
Stap 10: Gegevens loggen: Functie PatientFiles.m
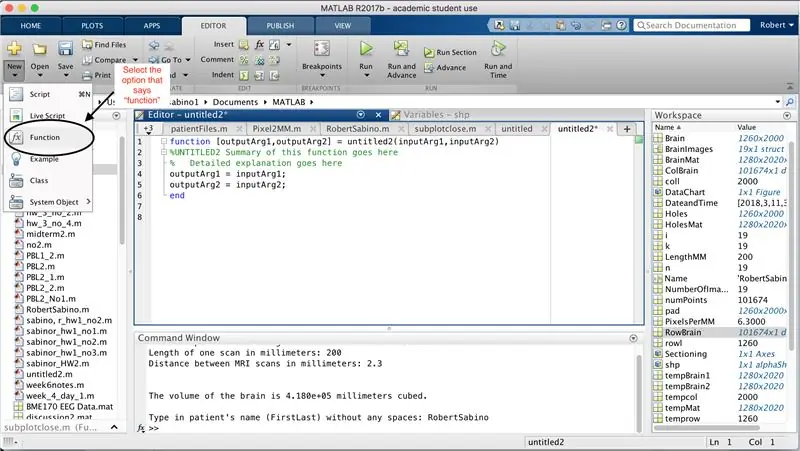
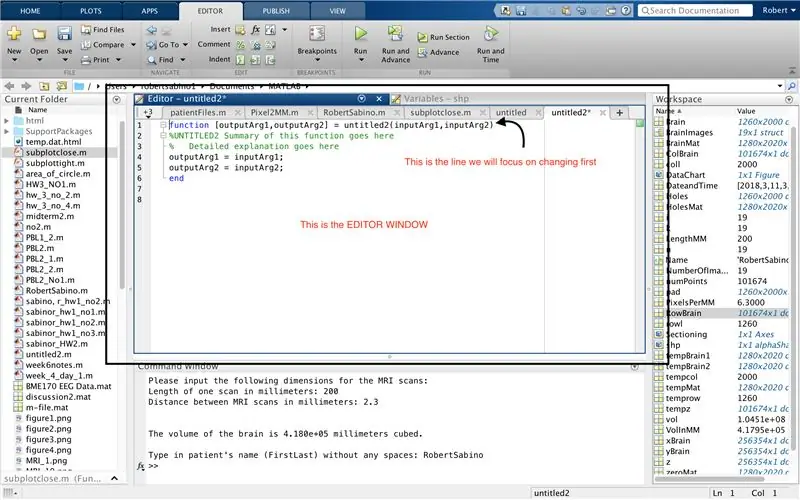

Net zoals bij het instellen van de laatste functie, klikt u op de knop in de linkerbovenhoek van het venster met de tekst "Nieuw" met een vet geel plusteken, en selecteert u de optie "functie", om in het editorvenster een spatie te openen die lijkt op die op de derde foto. Verwijder in de eerste regel de uitvoermatrix en vervang deze door alleen "output", vervang "untitled2" door "patientFiles", verwijder alle invoerargumenten en volg in plaats daarvan de opmaak die is gespecificeerd in de vierde afbeelding van de coderegel. De eerste regel van deze functie moet overeenkomen met de opmaak van de afbeelding.
Stap 11: Gegevens in bestanden loggen
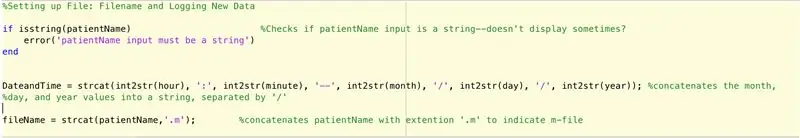

Om een bestand op te zetten om de gegevens te loggen die door de hoofdfunctie zijn gevonden (nog te beschrijven), moeten we deze stappen volgen (zoals voorgeschreven door de code regel voor regel).
- Controleer of de invoer voor patientName een string is.
- Als het geen tekenreeks is, geeft u weer dat de invoer van patiëntnaam een tekenreeks moet zijn.
- Beëindig de if-instructie (fout voorkomen).
- Stel een tekenreeksinstructie "DateandTime" in die het volgende formaat geeft: uur:minuten--maand/dag/jaar.
- Stel variabele bestandsnaam in op het volgende: patientName.m.
Nu naar het volgende gedeelte van de functie: Bestaat er al een bestand met deze naam?
1)Stel dat het bestand met deze naam al bestaat:
- Voer het bestand uit om de waarden uit het verleden in de wachtrij te krijgen
- Voeg de "DateandTime"-gegevens van de huidige iteratie toe als een nieuwe cel in de celreeks van x-waarden (index end+1)
- Voeg de huidige "brainVolume" -waarde toe als een nieuwe cel in de celreeks van y-waarden (index end+1)
- Sla de huidige variabelen op die in het bestand zijn geladen.
2)Stel dat het bestand met deze naam niet bestaat:
- Maak een nieuw bestand met de naam opgeslagen in de variabele "patientName"
- Voeg de huidige "DateandTime"-gegevens als een cel toe aan de lege celarray van x-waarden
- Voeg de huidige "brainVolume" -gegevens als een cel toe aan de lege celarray met y-waarden
- Sla de huidige variabelen op die in het bestand zijn geladen.
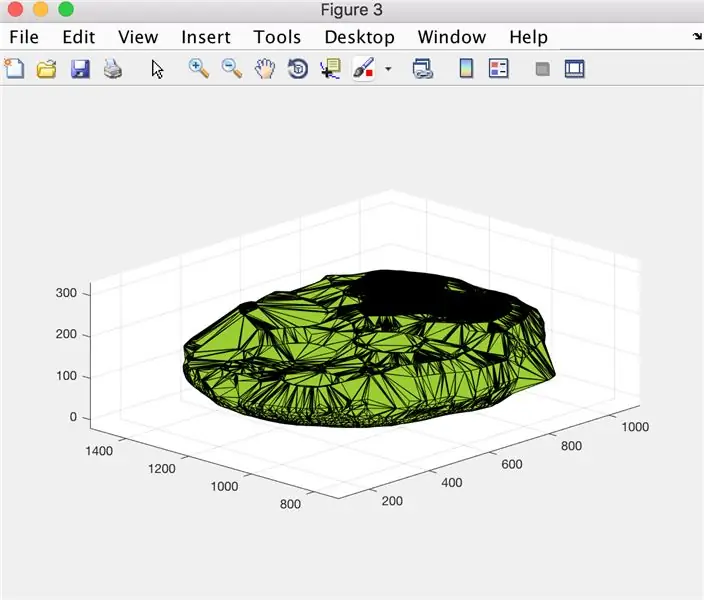
Stap 12: Gegevens loggen: een plot van hersenvolume weergeven in de loop van de tijd

- Converteer de x-waardenarray (xVals) naar een categorische array (xValsCategorical), om plotten mogelijk te maken
- Genereer figuurvenster 5
- Plot de punten aangeduid door "xValsCategorical" en "yVals" (met hersenvolume), met behulp van holle cirkels om punten aan te geven en te worden verbonden door stippellijnen
- Geef de plot een titel als: patientName Brain Volume Data
- Label de x-as zoals weergegeven in de afbeelding
- Label de y-as zoals weergegeven in de afbeelding
- Laat figuur 5 gelijk zijn aan output
Hieruit zal de functie patientName die wordt aangeroepen een bestand opleveren met bewerkte gegevens die het hersenvolume in de loop van de tijd bijhouden, en een plot die trends weergeeft.
Stap 13: hiaten in subplots dichten: Subplotclose.m

De functie, aangepast van code van https://www.briandalessandro.com, functioneert om de gaten te dichten tussen de subplotfiguren van de hoofdcode, wanneer de figuren die de MRI-beelden en de hersenlagen weergeven, worden gemaakt. De subplotfunctie die wordt gebruikt binnen subplotclose.m past de positie van de gegeven subplots aan zodat ze goed tegen elkaar aan passen in het aspect van de langere dimensie. Als de code bijvoorbeeld een matrix van 7 x 3 bedoelt, passen de rijen nauwsluitend omdat de rijafmetingen langer zijn. Als de code een matrix van 3 x 7 bedoelt, passen de kolommen precies, met gaten in de rijen, zoals weergegeven in de afbeeldingen van onze hoofdcode.
Stap 14: De hoofdcode: alles wissen en vragen om invoer

Om de hoofdcode te starten, klikt u op dezelfde knop met de tekst "Nieuw" in de linkerbovenhoek van het venster en selecteert u "Script" in plaats van "Functie" uit de eerdere secties. Typ de code zoals weergegeven in de afbeelding in het editorvenster. De coderegels doen de volgende taken in volgorde:
- Sluit alle geopende bestanden behalve 0, 1 en 2.
- Sluit alle figuurvensters.
- Wis alle variabelen in de werkruimte.
- Wis het opdrachtvenster.
- Weergave in het opdrachtvenster: Voer de volgende afmetingen in voor de MRI-scans:
- Vraag op een nieuwe regel in het opdrachtvenster: Lengte van één scan in millimeters:. Het antwoord van de gebruiker wordt ingesteld op de variabele "lengthMM".
- Vraag op een nieuwe regel: Afstand tussen MRI-scans in millimeters:. Het antwoord van de gebruiker wordt ingesteld op de variabele "ZStacks".
Stap 15: De hoofdcode: batchverwerking van de afbeeldingen

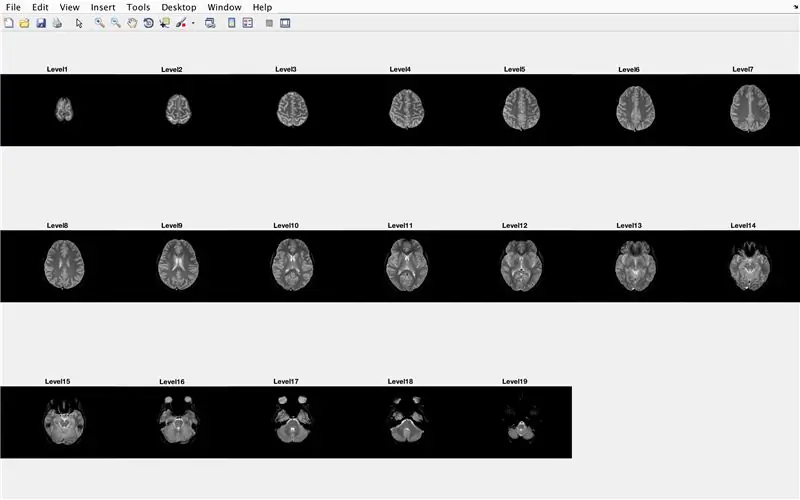
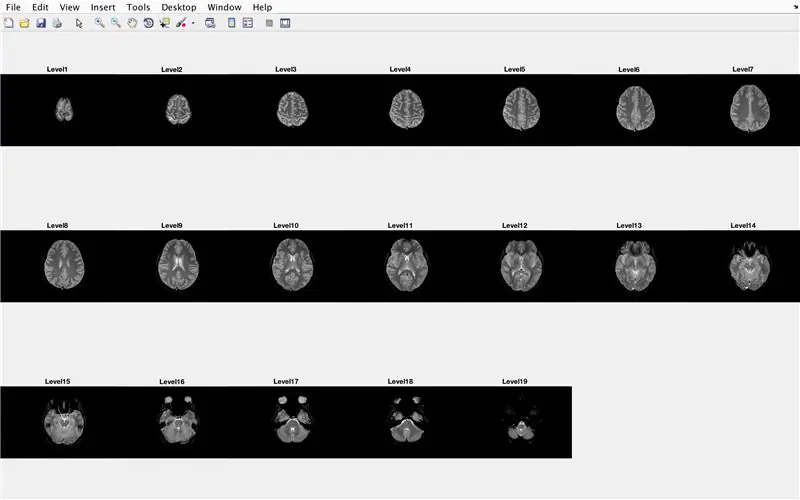
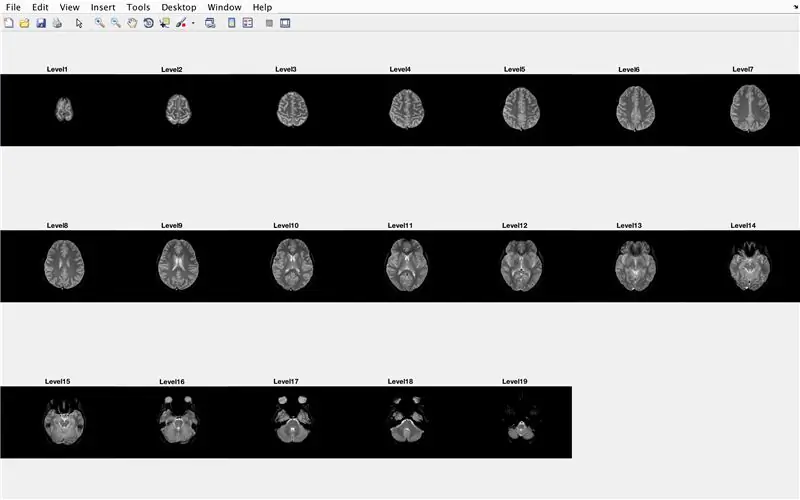
In deze sectie laadt de code de afbeeldingen (bestaande uit de MRI-scans van de dwarsdoorsneden van de hersenen) en slaat de namen van elk afbeeldingsbestand op in de variabele "Base" en geeft elk van de MRI-scans weer. Volg de code in de afbeelding, die het volgende doet:
- Maak een structuurarray "BrainImages" die informatie bevat over alle bestanden in de huidige map die passen in het naamformaat van MRI_().png
- Zet variabele "NumberofImages" gelijk aan het aantal elementen in de structure array "BrainImages"
- Open figuurvenster 1
- Stel een for-lus in om door te bladeren voor het aantal afbeeldingen dat in het bestand wordt geteld
- Voor elke lus is "CurrentImage" de respectieve naam van elk bestand MRI_i.png, met het iteratienummer als 'i'
- Genereer de subplot van 3 x 7 om de 19 afbeeldingen weer te geven die moeten worden geladen door "imshow"
- Geef elke afbeelding weer als een ander element in het subplot-figuurvenster
- Geef elk subplot-element een titel als Level_, waarbij blanco het iteratienummer van de for-lus is.
- Beëindig de for-lus (fout voorkomen)
Hierdoor worden in figuurvenster 1 alle MRI-scans in onbewerkte vorm weergegeven in een 3 x 7-configuratie zonder gaten in de x-oriëntatie.
Stap 16: De hoofdcode: opvulling

Met opvulling vermijden we het probleem van kleine verschillen in de afbeeldingsformaten die fouten kunnen opleveren voor niet-overeenkomende afmetingen in het geval dat de ene afbeelding iets groter is dan de andere.
- Open figuurvenster 2
- Laad de afbeeldingsmatrix van MRI_1-p.webp" />
- Zoek de grootte van de matrix van de afbeelding en stel in op "OriginalXPixels" (voor het aantal rijen) en "OriginalYPixels" (voor het aantal kolommen)
- Stel de matrix "BrainMat" in om uit allemaal nullen te bestaan met nog 20 rijen en 20 extra kolommen voor elk vlak, en 19 totale doorsneden, één per vlak.
- Stel "HolesMat" in om uit dezelfde driedimensionale reeks nullen te bestaan om de gatcoördinaten later in te voeren
- Maak "zeroMat" ter grootte van een pad plus twintig rijen en twintig kolommen, een tweedimensionale reeks nullen.
Stap 17: De hoofdcode: grenzen bepalen
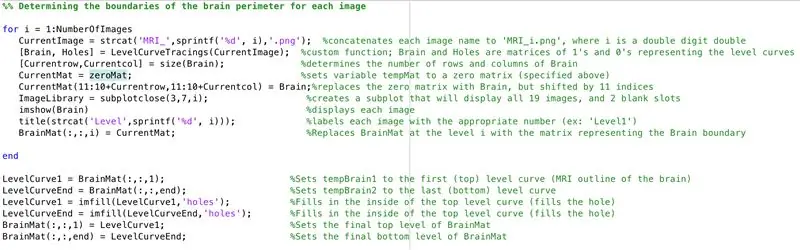
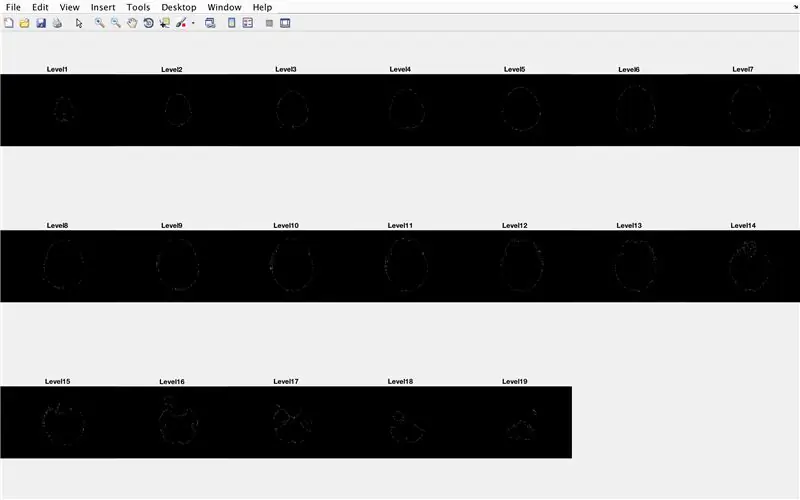
- Stel een for-lus in om de gegevens van elke eerder geladen afbeelding te doorlopen
- Op dezelfde manier van batchverwerking eerder, laadt "CurrentImage" bestanden op met "MRI_i.png", waarbij i het iteratienummer is
- Voer elke afbeelding door de verwerkingsfunctie "LevelCurveTracings2.m" die u eerder hebt gemaakt
- Zoek de grootte van de output "Brain", stel het aantal rijen in op "Currentrow" en het aantal kolommen op "Currentcolumns"
- Stel "CurrentMat" in op een matrix van nullen met de afmetingen gespecificeerd door "Currentrow" en "Currentcolumns"
- Centreer de gegevens van "Brain" in "CurrentMat", met een marge van 10 rijen aan alle kanten
- Genereer een subplot met afmetingen 3 x 7, om de grenzen van de afbeeldingen weer te geven
- Geef elk van de subplotelementen een titel in het figuurvenster
- Genereer driedimensionale matrix "BrainMat" bestaande uit elke grenslaag "CurrentMat"
- Beëindig de for-lus (om fouten te voorkomen)
De volgende subsectie vult de gaten aan de boven- en onderkant van de voorgestelde driedimensionale vorm in
- Stel "LevelCurve1" gelijk aan de eerste laag van "BrainMat" (onderkant van vast)
- Stel "LevelCurveEnd" gelijk aan de laatste laag van "BrainMat" (bovenkant van vast)
- Overschrijf "LevelCurve1" met een ingevulde laag
- Overschrijf "LevelCurveEnd" met een ingevulde laag
- Stel de ingevulde laag in als de onderste laag van "BrainMat"
- Stel de ingevulde laag in als de bovenste laag van "BrainMat"
Stap 18: De hoofdcode: de juiste Z-dimensie bepalen

De eerste drie regels bestaan uit het opzetten van een lege array "z", en het uitvoeren van eenvoudige conversiebewerkingen (pixels op lengte delen) om een juiste aflezing van het volume in mm^3 te krijgen
- Maak een for-lus om door elke laag te bladeren
- Vind het aantal enen in een bepaalde laag
- Converteer de z-coördinaten voor die naar waarden die zijn geschaald naar de juiste verhouding, ingesteld op "tempz", een kolomvector
- Voeg de z-waarde voor de niveaucurve toe aan de vector z
Hiermee worden z-coördinaten goed afgesteld.
Stap 19: De hoofdcode: X- en Y-coördinaten bepalen

Om nu de x- en y-posities van elk van de punten in de grenzen te bepalen.
- Initialiseer "xBrain" als een lege array
- Initialiseer "yBrain" als een lege array
- Stel een for-lus in om door elke geladen afbeelding te bladeren
- Stel een matrix met twee kolommen samen om de vlakke coördinaten van elk punt in de grens op te slaan, weergegeven door de kolomvectoren "RowBrain" en "ColumnBrain"
- Voeg "xBrain" toe aan de momenteel gevonden "RowBrain"-coördinaten
- Voeg "yBrain" toe aan de momenteel gevonden "ColumnBrain"-coördinaten
- Beëindig de for-lus (om fouten te voorkomen)
Stap 20: De hoofdcode: een driedimensionale structuur uitzetten, volume vinden en gegevens loggen
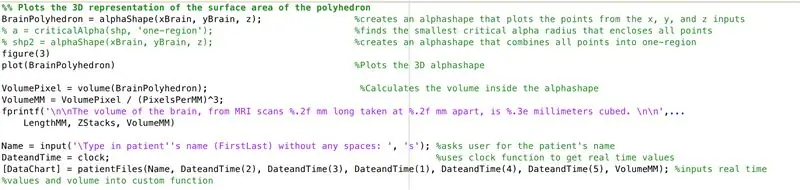

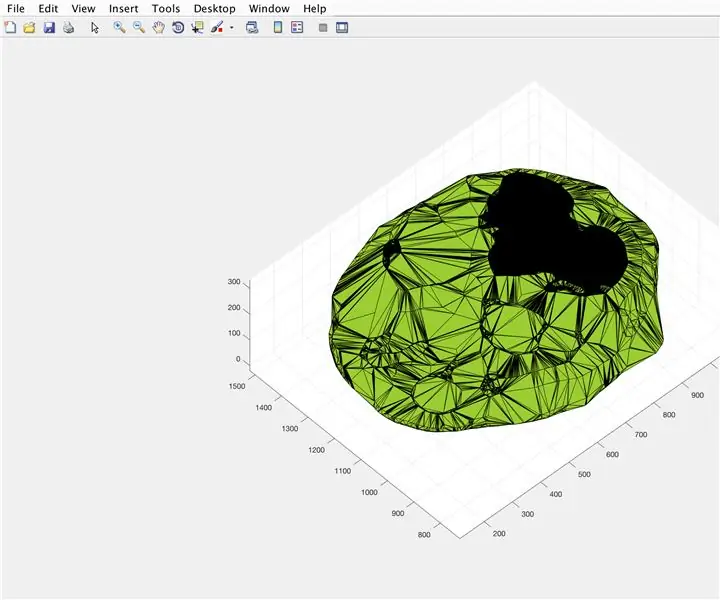
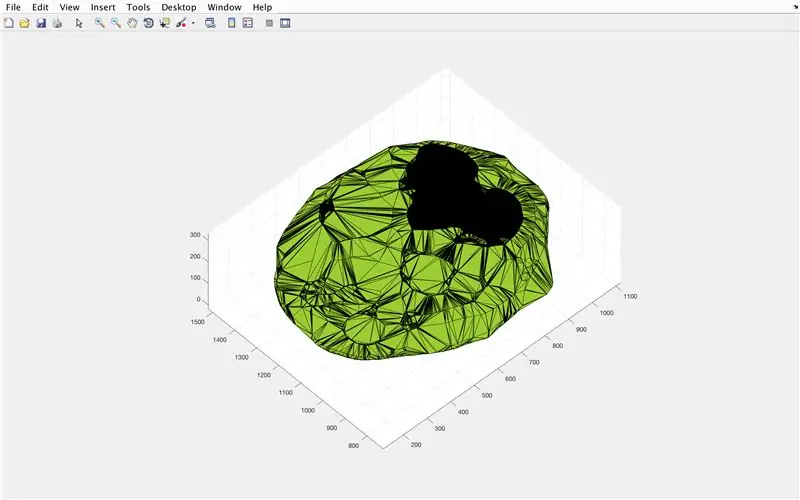
Met behulp van de functie alphaShape zullen we een driedimensionale structuur creëren waaruit we het volume van de hersenen kunnen berekenen.
- Gebruik de functie alphaShape, vul de vectoren "xBrain", "yBrain" en "z" in voor de x-, y- en z-coördinaten en stel deze in op "BrainPolyhedron"
- Open figuurvenster 3
- Plot de berekende alfavorm "BrainPolyhedron", weergave in het figuurvenster
- Bereken het volume van de alfavorm met behulp van een functie "volume" die werkt voor alfavormen
- Converteer het volume naar mm^3
- Druk het volume van de vaste stof af in het opdrachtvenster
- Vragen om een patiëntnaam in te stellen als invoer
- Krijg de huidige datum en tijd met de klok en stel in op "DateandTime"
- Roep de functie "patientFiles" op om de berekende gegevens te loggen en plotten
Vanaf hier zouden de tweede en derde afbeelding de figuren moeten tonen die verschijnen, en de vierde afbeelding die moet worden weergegeven in het opdrachtvenster.
Aanbevolen:
Tijd en datum ophalen van internet - zelfstudie: 9 stappen

Tijd en datum van internet halen - Zelfstudie: In deze zelfstudie leren we hoe u de datum en tijd van de NIST TIME-server kunt krijgen met behulp van M5Stack StickC en Visuino, Bekijk een demonstratievideo
Automatisch volgen van lichtbronnen: 5 stappen
Automatisch volgen van lichtbronnen: in deze les zullen we een servomotor, een fotoresistor en een pull-down-weerstand gebruiken om een automatisch volgend lichtbronsysteem te assembleren
Een fitnesshorloge dat de groei van bacteriën kan volgen: 14 stappen

Een fitnesshorloge dat de groei van bacteriën kan volgen: Bacteriën spelen een belangrijke rol in ons leven. Ze kunnen heilzaam zijn en ons medicijnen, bier, voedselingrediënten enz. opleveren. Het continu monitoren van de groeifase en de concentratie van bacteriële cellen is een cruciaal proces. Dit is een belangrijke ro
Het hoofd van meneer Wallplate draait zich om u te volgen: 9 stappen (met afbeeldingen)

Het hoofd van Mr. Wallplate draait om u te volgen: Dit is een meer geavanceerde versie van Mr. Wallplate's Eye Illusion Robot https://www.instructables.com/id/Mr-Wallplates-Eye-Illusion. Met een ultrasone sensor kan het hoofd van meneer Wallplate u volgen terwijl u voor hem loopt. Het proces kan worden samengevat
Rechte kopteksten omzetten in haakse kopteksten (in een mum van tijd): 4 stappen

Converteer rechte headers naar rechthoekige headers (in een mum van tijd): na het zien van de aankondiging voor de arduino-wedstrijd, zei ik, hey waarom niet proberen. uit en kreeg de barebones arduino-kit, met de bedoeling "het op mijn manier te maken". Een van die veranderingen was een van de eerste dingen die je
